当然是躺在家里啥也不干了!
不过,随着各地已经开始复工复学,大家可能很难一直躺在家里为国家做贡献了。
而我们也不是医卫人员,更不是科研人员,作为一名普通民众,有没有什么办法可以为这场病毒与人类的战役出一份力呢?
“既然你诚心诚意的发问了,那我就大发慈悲地告诉你!”

其实还真有办法,那就是加入Folding@Home项目,然后下载他们的计算软件在自家电脑上运行,利用自己电脑闲置的GPU算力来帮助科学家模拟新型冠状病毒的蛋白质折叠过程。
看到这,小问号,你是否有很多朋友?为啥我下个软件在电脑上运行就能帮助到科学家了?
别急,首先我们来了解下Folding@Home是干什么的。
这个组织是由斯坦福大学化学系的潘德小组(Pande Group)所开发创建的一个研究蛋白质折叠、蛋白质错误折叠或聚合以及由此引起的相关疾病的分布式计算工程。也是目前吉尼斯认定的世界最大的分布式计算项目。

分布式计算简单来说就是在许多台电脑上同时运行计算,而且最后还必须出一个结果。自2000年创建至今,已经有超过百万人参与项目并提交了运算结果,它的计算能力总和也达到了全球计算机TOP10水平。
而如此恐怖的计算能力其实只为了弄明白一个问题:蛋白质是怎么折叠的?
在生物学中,蛋白质是由氨基酸以“脱水缩合”的方式组成的多肽链经过盘曲折叠而形成的具有一定空间结构的物质。
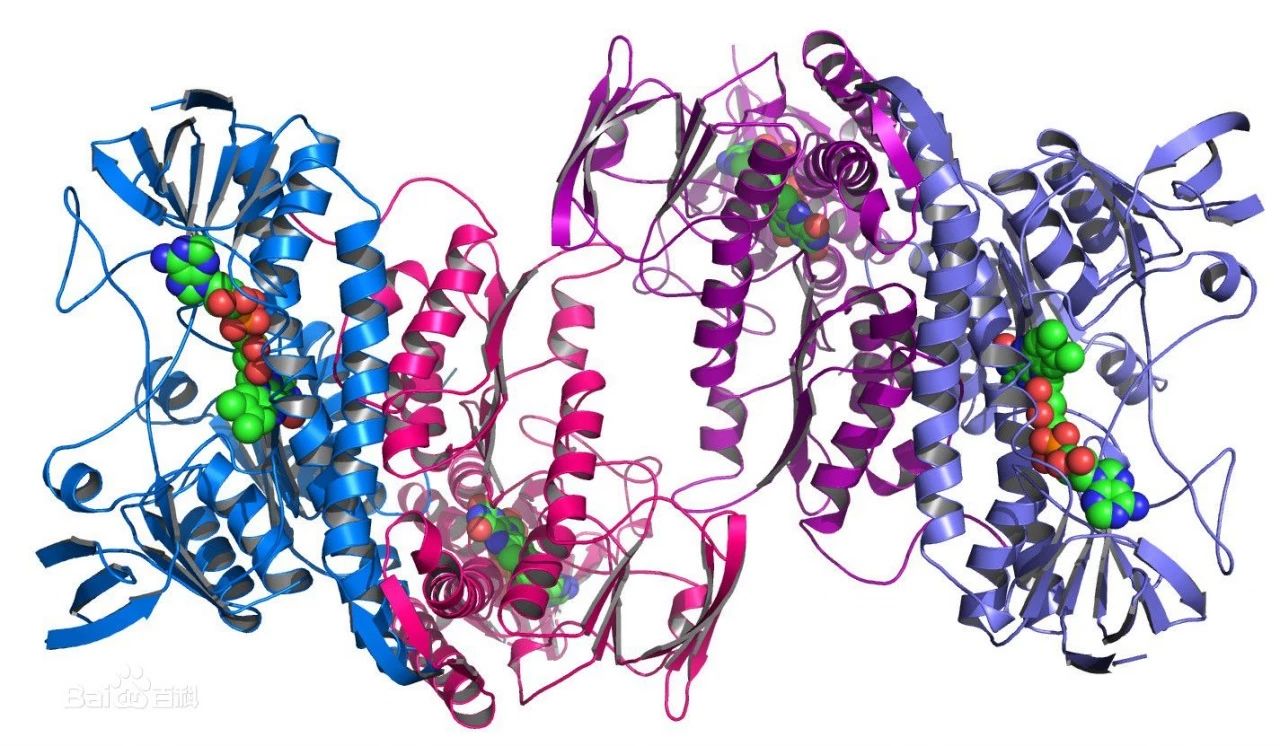
它可以作为酶,来促进生物体内各项生化反应的完成;也可以作为抗体,用来识别来自外界的有害成分并允许免疫系统攻击这些“入侵者”;此外,它还是我们骨骼、肌肉、头发、皮肤和血管等组织的主要组成成分。
鉴于蛋白质对生物的作用如此重要,科学家们对人类所有基因组都进行了测序(生物学中所有蛋白质的蓝图),但对于蛋白质来说,仅仅知道基因组序列并不能使我们充分了解蛋白质的功能,更无法知道它是如何工作的。
因为结构决定其功能。
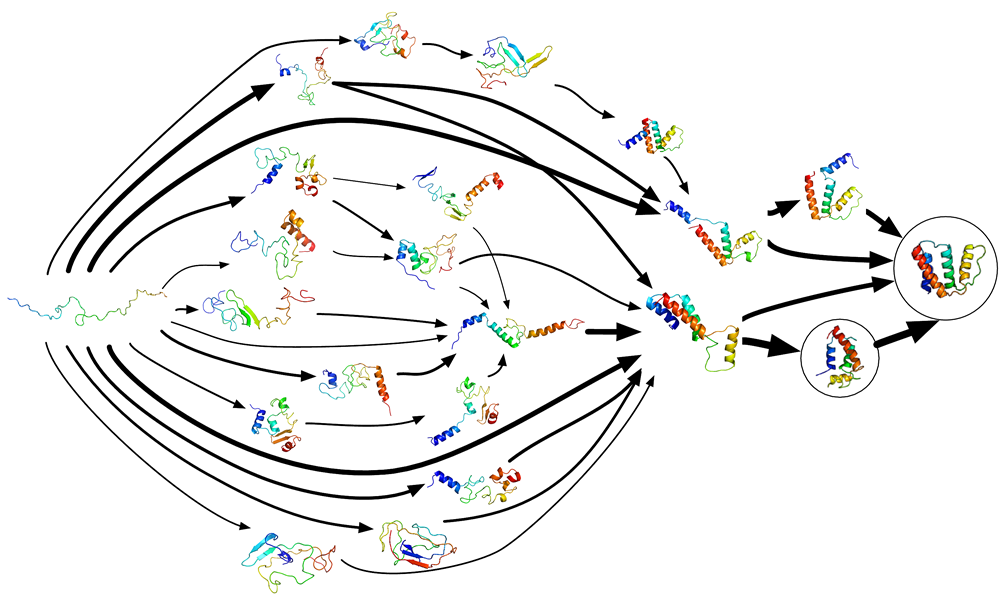
蛋白质可能的折叠过程
为了执行某些特定功能(比如,作为酶或抗体),它们必须变成特定形状,因此蛋白质会在细胞环境(特定酸碱度、温度等)下自己把自己组装了(活生生把自己玩成了乐高),这种自我组装的过程,也被称为蛋白质折叠。
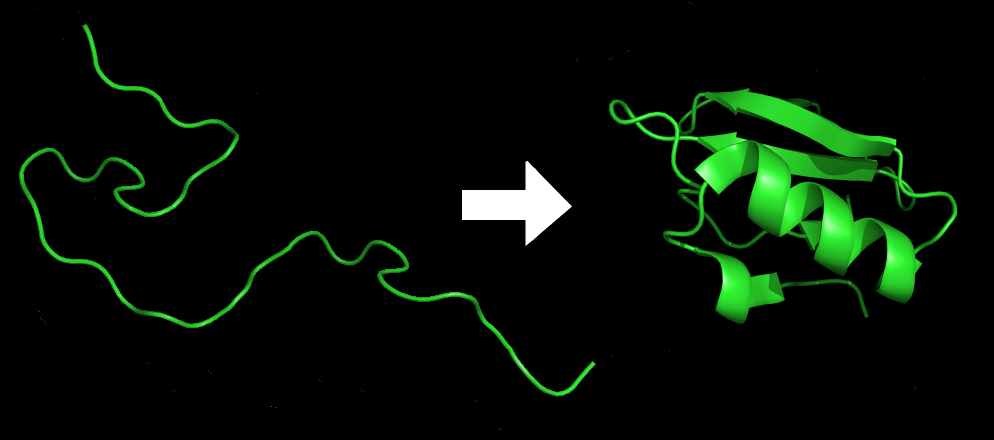
蛋白质从一个不稳定的线条折叠成原生状态
可尴尬的问题是,蛋白质可以在极短的时间内从一级结构折叠成立体三维结构,研究者却无法捕捉到这个过程,甚至都不能在短时间内通过氨基酸序列计算出蛋白质结构。
在这种情况下,Floding@Home项目应运而生,它们的使命正是通过计算机模拟出蛋白质折叠过程,并从中寻找致病机理。
而引起此次新冠疫情的病毒SARS-COV-2(注意哈,不是非典的那个SARS),在它表面存在着许多“S刺突蛋白”,这种蛋白可以附着在肺泡细胞表面,从而让人体出现严重的急性呼吸系统综合征。
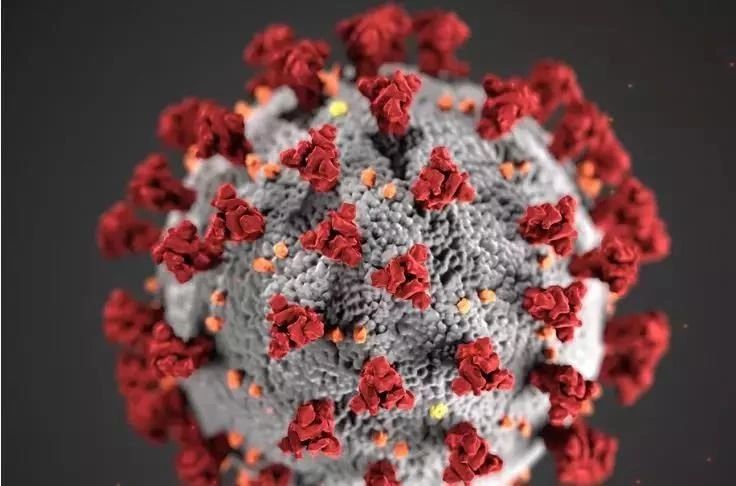
因此目前许多针对性研究都聚焦于这种刺突蛋白上。
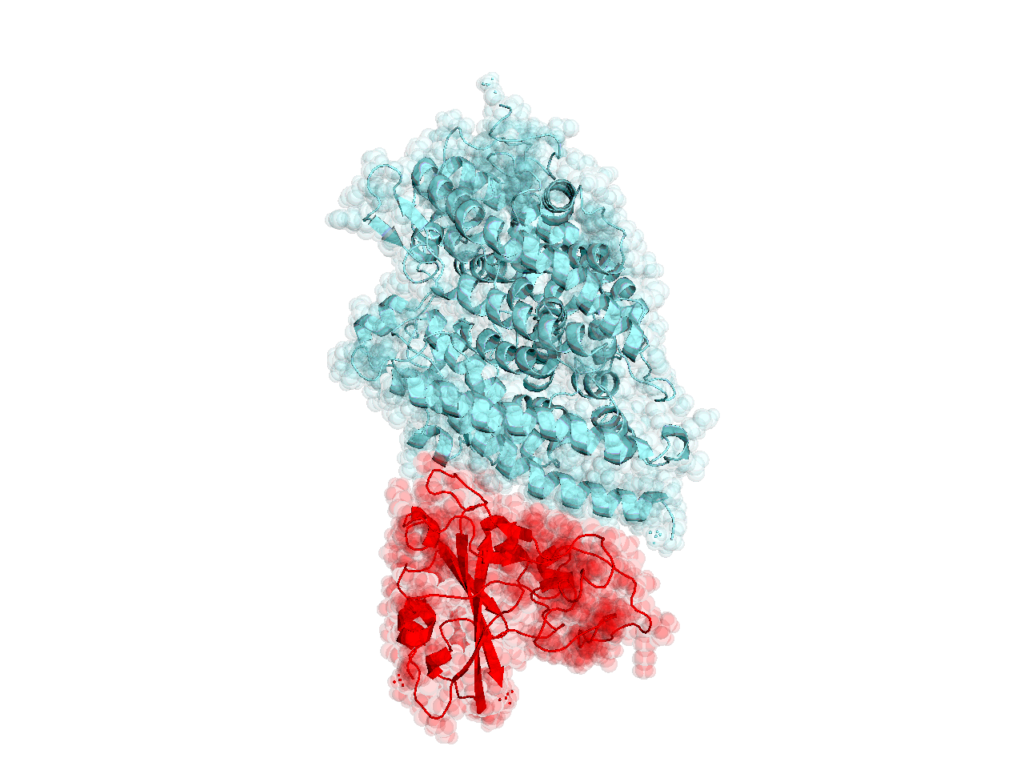
不过,科学家目前尚不完全了解刺突蛋白的工作原理(功能)。于是Floding@Home联合凯特琳癌症治疗及研究中心开展了“模拟新型冠状病毒蛋白质折叠”的科研项目。

然后他们把项目上传到了boinc平台,可能有同学不知道这又是个啥,来给大家简单介绍一下。
Boinc平台是美国加州大学伯克利分校的一个团队所开发的志愿计算平台。

它的主要作用是为大多数科研组织提供超强的运算能力,因为在科学研究中,有很多复杂的问题需要使用计算机的计算,并且计算量还不是一般的计算机可以完成的。
但这些做科研的组织中,其实大部分属于学校旗下或者非商业的实验室,哪有钱去采购如此大算力的计算机(毕竟像天河二号这种级别的计算机可不是每个大学都拥有的)。

所以boinc的出现恰好解决了这个问题。通过BOINC,研究组织可以发布任务给无数个志愿者,每个志愿者贡献自己计算机的一点GPU算力进行运算,最后把结果传回研究组织的手里。
而我们借助这款软件,就可以利用电脑闲置的GPU来协助运算蛋白质模拟背后庞大的数据,助力这次新冠肺炎的研究。
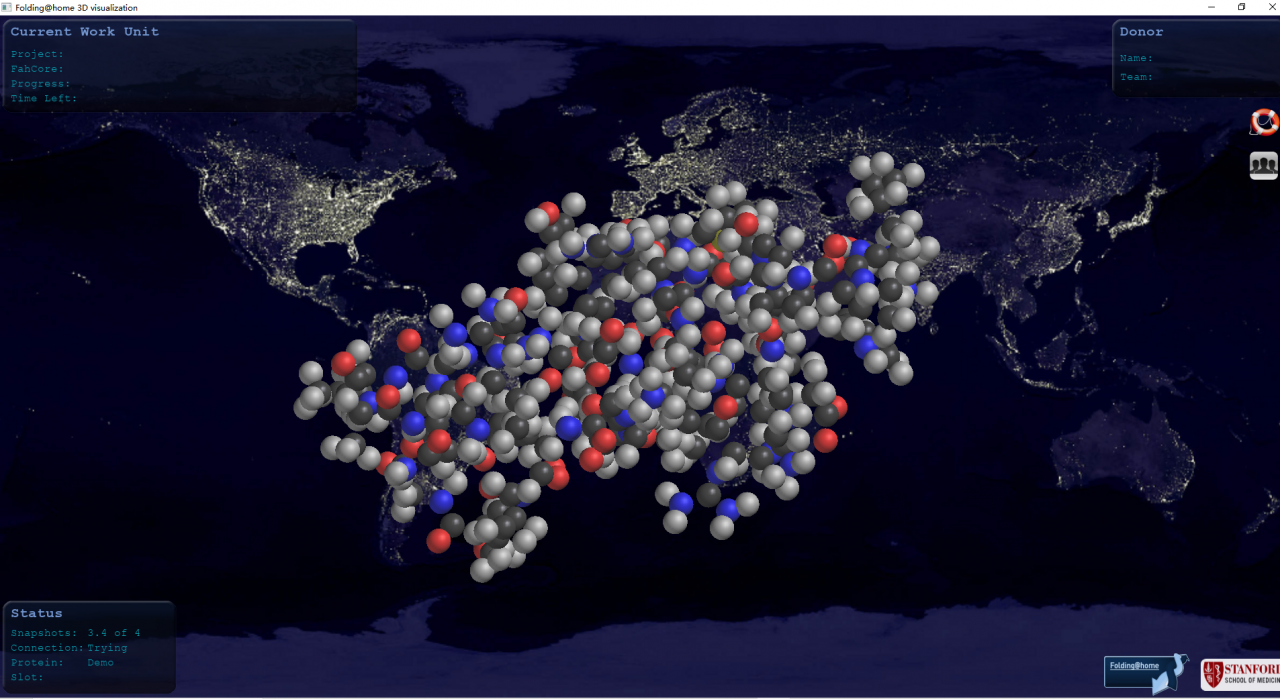
小编在自己电脑上运行的Folding@Home软件
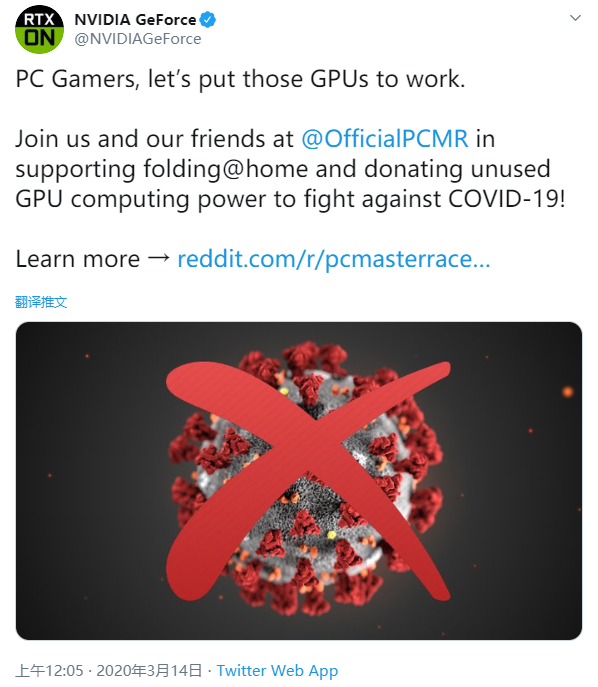
另外,著名的显卡制造商NVIDIA也在推特上呼吁玩家捐赠出闲置的GPU算力,支持对抗新冠病毒的项目Folding@Home。
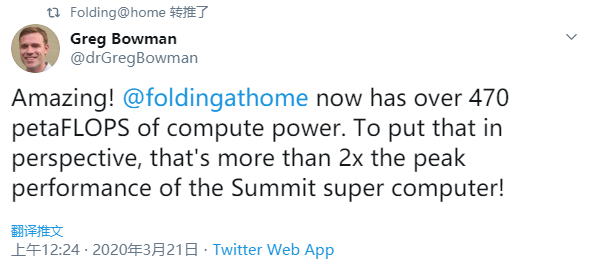
在英伟达呼吁玩家之前,全球有约3万人参与到了Folding@home的SARS-COV-2运算中,当英伟达为项目发声后,截止目前参与的玩家数激增至约40万人,该项目的算力也由先前的100 petaFLOPS(单位为每秒千万亿次浮点运算),暴增至470 petaFLOPS(每秒47亿亿次浮点运算),是目前TOP 500超算冠军Summit的峰值性能200 petaFLOPS的2倍还多。
话说回来,如果老黄可以给我发个2080ti,我可以7x24小时给你跑,不管怎么样,都是为了LOVE&PEACE嘛!







 扫码下载APP
扫码下载APP

 科普中国APP
科普中国APP
 科普中国
科普中国
 科普中国
科普中国