格特隐球菌(Cyptococcus gattii)属于新生隐球菌复合体(Cryptococcus neoformans complex),既往被认为是新生隐球菌复合体的变种(C. neoformans var. gattii),但最终被确立为独立的物种[1]。与新生隐球菌不同,格特隐球菌多感染免疫力正常人群[2]。格特隐球菌以担孢子和酵母细胞形态被吸入,引起人或动物肺部感染,再传播到中枢神经系统,导致脑膜炎或脑膜脑炎,感染后具有致命性;除了中枢神经系统和肺部,还可累及皮肤、骨、关节、喉淋巴结、腹腔、前列腺、眼睛、尿路和循环系统等部位[3]。
格特隐球菌主要分为4个谱系:VGⅠ、VGⅡ、VGⅢ和VGⅣ[2,4],其中VGⅡ是最古老的谱系,其次是VGⅣ[5]。通过对临床、动物和环境分离株的分析发现[2,6-9]:在所有分离株中,最常见的分子类型为VGⅡ,占所有分离株的47%,其次是VGⅠ(34%)、VGⅢ(11%)和VGⅣ(8%);在临床分离株中,VGⅠ和VGⅡ具有相似的比例,VGⅢ和VGⅣ次之;而大多数环境分离株属于分子型VGⅡ,其次是VGⅠ和VGⅢ,环境分离株则很少出现VGⅣ型。
格特隐球菌感染主要分布于热带和亚热带地区。1999年在地处温带的加拿大温哥华岛[6]及美国西北部地区[10]发生了格特隐球菌暴发流行,我国及欧洲地区亦有病例报道[11-13]。近期研究显示,其所致感染病例地理分布正在不断扩大[14]。目前格特隐球菌的致病机制、毒力作用机制、暴发流行和进化机制尚不完全明确,致使安全有效的治疗药物和疫苗开发处于停滞状态。随着测序技术的不断发展,针对格特隐球菌的基因组学分析将有助于解决这一难题。因此,本文就格特隐球菌的基因组测序和基本特征、基因组进化、重要毒力基因以及比较基因组研究予以概述。
1 基因组测序及其基本特征
格特隐球菌为二倍体,基因组包含14条线性染色体。随着测序技术的不断发展,虽然越来越多的格特隐球菌菌株被测序,但由于真菌基因组具有较多的重复序列,目前仅有一株VGⅠ菌株(WM276)和一株VGⅡ菌株(R265)基因组拼接至染色体水平。
VGⅠ型的WM276由来自加拿大不列颠哥伦比亚大学迈克尔·史密斯实验室的J. Kronstad等于2011年测序完成,基因组组装基于BAC文库构建的物理图谱和包含来自BAC克隆末端的测序reads支持完成。WM276基因组长约18.4 Mb,包含8个gap区域,8条染色体的两端鉴定出由基序重复序列组成的端粒序列(TTAGGGG)n, 4条染色体的一端鉴定出端粒序列,其余2条染色体两端均未鉴定出端粒序列。其中,最大的染色体2.23 Mb,最小的染色体0.52 Mb,平均G+C含量47.9%,共计编码约6560个基因[15]。
VGⅡ型的R265由Broad研究所测序完成,是Broad真菌基因组计划的一部分,该菌株是引起加拿大温哥华地区格特隐球菌感染的爆发菌株。作为NHGRI白皮书的一部分,Broad研究所还对其他格特隐球菌菌株进行了测序,以辅助提高R265的基因组组装水平。R265基因组约17.5 Mb,G+C含量47.8%,最大的染色体2.21 Mb,最小的染色体0.52 Mb,共编码约6470个基因。此外,研究人员还拼接得到了0.03 Mb,G+C含量33.4%的线粒体基因组[15]。
VGⅢ和VGⅣ型别的菌株尚无基因组相对完整的代表菌株。
2 基因组进化特征
早期研究中,格特隐球菌的进化分析主要基于少数基因位点。2008年,Bovers等[5]通过对6个基因位点LAC1、ITS1/2、IGS、RPB1、RPB2和TEF1的分析,鉴定VGⅡ为格特隐球菌菌株的起源,其次是VGⅣ,同时VGⅣ又为姐妹分支VGⅠ和VGⅢ的起源。除6个基因位点之外,通过对线粒体基因组上两个基因位点mtlrRNA和ATP6的分析发现,格特隐球菌不同谱系之间在线粒体基因组上存在较为广泛的基因重组[16]。利用PCR指纹图谱对4个基因座ACT1、IDE1、PLB1和URA5的分析,进一步确认VGⅡ为格特隐球菌最古老的谱系,且4大谱系包含B、C两个血清型[17-18]。对这4个基因座的进一步分析显示,格特隐球菌较新生隐球菌位于进化的下级。此外,基于10个基因位点ACT1、LAC1、IDE1、ITS1/2、IGS、PLB1、RPB1、RPB2、TEF1和URA5,并通过对进化时间的推算,进一步确立了VGⅡ的祖先地位[19],VGⅡ与其他分支的分离约在6200万年前(范围:3300~8700万年前),VGⅥ分支与VGⅢ和VGⅠ分支的分离在5200万年前(范围:2700~7700万年前),VGⅢ和VGⅠ的分离约在4700万年前(范围:2600~6700万年前)(图1)[2]。
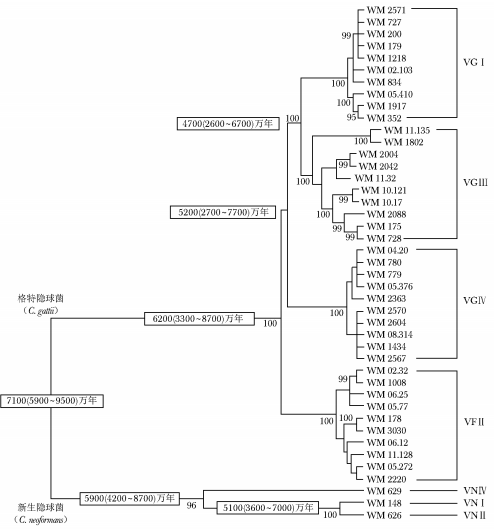
图 1格特隐球菌菌株多位点序列分型进化树[2]
近年来,随着测序技术的不断发展,全基因组序列分型(whole-genome sequence typing,WGST)得到越来越广泛的应用。2011年,Gillece等[20]对太平洋西部高毒力VGⅡ型菌株的传播分析发现,VGⅡ内部具有3种不同亚型的菌株 (VGⅡa、VGⅡb和VGⅡc),其中VGⅡa遗传多样性最高,其次是VGⅡc和VGⅡb,而VGⅡc基因型菌株是引起太平洋西部感染最常见的菌株,在基因组上与温哥华岛爆发亚型VGⅡa和VGⅡb具有较大差异。基于多位点序列分型和多位点微卫星分型分析也具有相似的发现,并且进一步得到WGST的证实[21-23]。目前所有研究均表明,VGⅡ3个亚型的出现是基因组重组的结果,且重组发生在进入太平洋西部之前。
此外,VGⅠ、VGⅡ、VGⅢ和VGⅣ在不同地区的分布具有地区特异性:(1)美洲:主要为VGⅡ和VGⅢ型,其中大多数VGⅡ临床株来自于南美洲[7]。VGⅡ的两种主要亚型VGⅡa和VGⅡb主要来自加拿大和太平洋西北地区[6]。此外,新的高毒力亚型VGⅡc也已在俄勒冈州以及太平洋西北地区被鉴定出来[24]。(2)欧洲:仅有VGⅠ和VGⅡ的报道,且多数病例为VGⅠ感染[25]。(3)非洲:虽然对非洲的格特隐球菌知之甚少,但其流行病学似乎与世界上其他地区大不相同,主要是VGⅣ类型菌株,且主要来自非洲南部[26]。(4)亚洲和澳大利亚:主要的分子类型是VGⅠ,其次是VGⅡ,VGⅢ很少被分离出来[27]。
3 重要毒力因子
研究表明,荚膜、黑色素、生理温度下的生长能力、降解酶以及交配型等与隐球菌致病性紧密相关[28-29]。
3.1荚膜
荚膜由90%~95%的葡萄糖醛酰木糖基甘露聚糖( glucuronoxylomannan,GXM)、5%的半乳糖木糖甘露聚糖和小于1%的甘露糖蛋白构成,对于隐球菌在其宿主中的存活至关重要,其通过为酵母细胞提供直接保护以增加隐球菌的适应性。例如,在不存在噬菌素的情况下,荚膜抑制噬细胞对隐球菌的吞噬作用[30],且抵抗吞噬体消化[31]。荚膜多糖亦直接作用于宿主,在巨噬细胞中,隐球菌将多糖从其荚膜中释放到吞噬体周围的囊泡中,且这些囊泡在宿主细胞的细胞质中积累导致巨噬细胞功能障碍和溶解[31-32]。
目前已报道与荚膜生成相关的基因及其功能包括CAP59(跨膜蛋白,介导GXM的运输)、CAP64(与荚膜生成和宿主致命感染相关)、CAP60/CAP10(编码定位于核膜和细胞质的蛋白)、CAS1/CAS3(介导GXM乙酰化)、UXS1/UGD1/CAS31/CAS32/CAS33/CAS34/CAS35(介导GXM的木糖基化)[29]。
3.2黑色素
黑色素是一种带负电荷的疏水性高分子量颜料,由酚类化合物的氧化聚合形成[33]。在某些邻二酚化合物如3,4-二羟基苯丙氨酸[34]的存在下,漆酶催化隐球菌中的黑色素合成。研究发现,从人脑组织中回收的新生隐球菌细胞是黑化的,产生黑色素的新生隐球菌毒性更高[33]。此外,与黑化的新生隐球菌细胞相比,非黑化细胞对氧化剂的敏感性较高,且易被抗真菌药物(如卡泊芬净和两性霉素B)杀死[35]。由于吞噬作用后氧化剂的产生是免疫细胞介导抗微生物作用的重要机制,因此这些结果表明黑色素可通过保护真菌细胞免受免疫系统的攻击来增强毒力。此外,观察到的黑化新生隐球菌能更好地抵御吞噬作用和抑菌作用,也进一步证实了这一点。
两种漆酶基因LAC1和LAC2被鉴定为黑色素生物合成的中心酶[29]。其他基因,包括VPH1、CLC1、CCC2、ATX1和MBF1也被发现是黑色素生成所必需的。
3.3生理温度下的生长能力
在生理温度下的生长能力对于新生隐球菌和格特隐球菌的毒力是必不可少的。虽然一些隐球菌物种(例如播散隐球菌)也具有胶囊,并产生黑色素,但很少能在37℃下生长,因此不会在哺乳动物中引起感染[36]。隐球菌在鸟粪富集,但鸟类不会被感染,可能是因为隐球菌在40~42 ℃的禽类体温下不能很好地生存。因此,这种温度限制是隐球菌致病性的重要决定因素。
早期研究发现了十几种高温生长所必需的基因,包括CNA1、CNB1、CPA1、CCN1、TPS1、TPS2、MGA2、RAS1、SOD2、TSA1、ILV2、SPE3/LYS9、MPK1和STE20等[36]。
3.4降解酶
3.4.1蛋白酶
降解包括胶原、弹性蛋白、纤维蛋白、免疫球蛋白和补充因子在内的宿主蛋白,引起宿主组织破坏,为隐球菌提供营养物质[31]。
3.4.2磷脂酶
能够水解甘油磷脂中的一个或多个酯键,可破坏膜稳定性,导致细胞裂解和脂质第二信使的释放[37]。隐球菌分泌的磷脂酶表现出磷脂酶B、溶血磷脂酶水解酶和溶血磷脂酶转酰酶活性。与蛋白酶相同,磷脂酶有助于宿主细胞膜的降解,从而促进细胞裂解。在用于感染小鼠的菌株中,磷脂酶表达与毒力呈剂量依赖性相关[37]。PLB1基因的破坏导致体内毒力降低,抑制巨噬细胞样细胞系中的生长[38]。磷脂酶还可裂解二棕榈酰磷脂酰胆碱,即一种肺表面活性剂主要成分,从而有助于真菌扩散[39]。最近研究表明,新生隐球菌的磷脂酶B增强了新生隐球菌对人肺上皮细胞系的黏附[40]和小鼠模型中隐球菌病的传播[41]。
3.4.3脲酶
催化尿素水解成氨和氨基甲酸酯,并且是某些致病菌的重要致病因子[39]。隐球菌脲酶(编码基因:URE1)是重要的毒力因子,用URE1突变株感染的小鼠比用野生型菌株H99感染的小鼠寿命更长[42]。虽然脲酶不是大脑生长所必需的,但静脉接种URE1突变株后,脑、脾和其他器官的传播模式与野生型菌株显著不同,因此提出URE1通过增强微毛细管内的酵母螯合作用对中枢神经系统入侵发挥作用。
3.5交配型
一些有趣的观察结果表明,交配型也是一种毒力因子。首先,MATα细胞比MATa细胞更普遍。例如,MATα交配型比MATa在环境分离株中的含量高40倍,在临床分离株中的含量高30倍[43]。其次,当在隐球菌病的小鼠模型中研究血清型D(除交配型基因座外其他基因相同)菌株JEC21时,发现MATα菌株比MATa菌株毒性更高[44]。MATα细胞比MATa细胞更普遍和更具毒性的发现促进了对MATa交配型基因座的分析。最初,仅在MATa菌株中约50 kb的区域被定义为MATα基因座,且其包含许多α特异性基因,包括STE12a[45]。然而,MAT基因座的实际大小似乎远大于此,在新生隐球菌和格特隐球菌中的长度均超过100 kb,含有>20个基因,包括那些参与信息素产生、传感、建立细胞类型特性、MAP激酶途径的成分等[46]。
此外,一些基因参与隐球菌多个致病相关过程,在隐球菌致病过程中发挥重要作用(表1)。
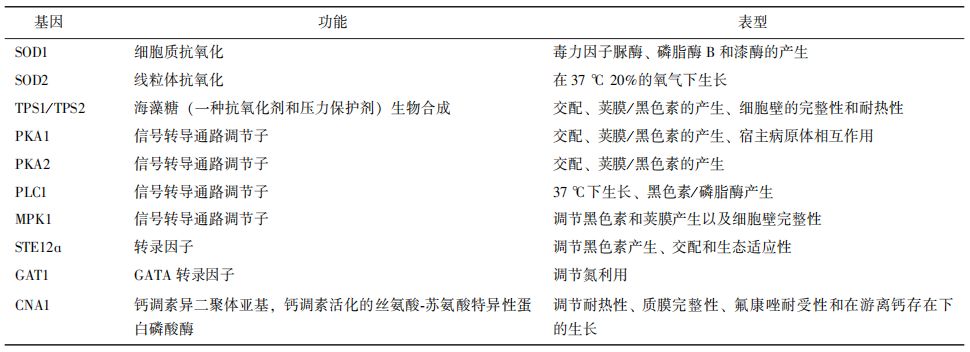
表1 格特隐球菌重要致病性相关基因[2]
4 比较基因组学研究
不同谱系的格特隐球菌菌株基因组长度(17.32~18.36 Mb)和基因个数(6456~6763)差别不大,但谱系之间存在较大的遗传变异(平均基因组一致性93%,52 SNPs/kb),而谱系内部菌株平均基因组一致性为97%,每kb少于6个SNPs[47]。格特隐球菌菌株的第一个全基因组比较分析由DSouza 等[15]于2011年完成,主要对单个VGⅠ分离株WM276(代表导致全球大多数格特隐球菌感染的分子类型)和VGⅡ分离株R265(代表高毒力VGⅡa温哥华岛爆发基因型)进行了分析。随着测序和分析技术的不断发展,研究人员逐渐关注到格特隐球菌各个谱系的菌株。
4.1基因组结构
研究表明,4个谱系菌株染色体结构高度保守,其中VGⅡ尤为保守[47]。几乎所有的同线性变异都在3个密切相关的谱系VGⅠ、VGⅢ和VGⅣ中。进一步研究发现,大于100 kb的染色体重排约15个,平均而言,约2.6%的区域发生了重排。15个重排区域包括10个易位(7条染色体间和3条染色体内)和5个支架融合,其中大多数(15个中的13个)与在着丝粒中发现的TcN转座子簇相关[48],表明这些主要是全染色体臂重排。对更多的菌株基因组的研究进一步证实了存在4种染色体重排,包括VGⅡ特有的染色体融合、VGⅢ独有的两种易位(分别为700 kb和140 kb)以及VGⅣ特有的一种易位(450 kb)。由于一些重排事件会产生缺失的染色体区域或其他非整倍体和不可存活的后代,因此这些变化可能影响谱系之间的遗传交换能力。
除了染色体重排,VGⅡ和VGⅢ谱系菌株中还发现存在非整倍体[47]。如对VGⅡ谱系动物分离株B8828的研究发现,scaffold 13拥有额外两个拷贝;对VGⅢ谱系临床株CA1280的研究发现,scaffold 2二倍体缺失;对VGⅡ临床分离株LA55研究发现,scaffold 1中具有60 kb的染色体内重复。已有研究显示,染色体拷贝数的变异影响新生隐球菌的毒力[49],且可通过增加唑类药物靶标(ERG11)或转运蛋白(AFR1)的拷贝数进一步提高对唑类药物的抗性。然而,格特隐球菌分离株中的这类基因似乎没有更高的拷贝数,表明这些非整倍体与已知的抗药性机制无关,可能对这些分离株产生其他影响。
对线粒体基因组结构的分析发现,线粒体基因组谱系之间的重组比染色体的重组更加频繁。研究发现,VGⅠ谱系菌株线粒体基因组序列与VGⅡ的相似性远高于核基因组,且对两两谱系Fst值[50]的计算也表明线粒体基因组[VGⅠ-VGⅢ (0.642), VGⅠ-VGⅣ (0.672), VGⅢ-VGⅣ (0.658)]比核基因组[VGⅠ-VGⅢ (0.885), VGⅠ-VGⅣ (0.894), VGⅢ-VGⅣ (0.885)]重组率更高[49]。
4.2特异基因
Farrer等[46]涉及4个谱系16株格特隐球菌菌株的研究发现了737个谱系特异或者在2或3个谱系特异的直系同源基因簇(表2)。这些基因主要来源于许多小的染色体内部变化,并在所有染色体散在分布。研究人员进一步对这些基因Pfam结构域和GO的功能富集进行分析,发现每个谱系携带一类独特的基因功能子集,这些基因可能参与毒力和疾病预后,包括Fe+/Cu+结合基因,维持或影响线粒体形态的基因,以及应激反应相关基因。其中,最大GO富集类别为氧化应激。
VGⅠ具有携带铁还原酶样跨膜组分和铁还原酶NAD结合结构域基因的特异扩增。铁还原酶参与毒力因子黑色素产生和对唑类抗真菌药物的抗性[51]。总体而言,VGⅠ具有4个谱系中最少的谱系特异基因,但有12个显著富集的Pfam结构域,其中包括被认为参与DNA修复的HORMA结构域的基因扩增[52]。
VGⅡ分离株携带的特异基因包括涉及膜转运相关的膜分泌载体膜蛋白、Prmt1(Fop)染色质相关蛋白[53]以及在伴侣蛋白中发现的热休克蛋白70(heat shock protein 70,HSP70)。在新型隐球菌中,对HSP70基因家族成员Ssa1的缺失表明,HSP70作为真菌毒力所需的应激相关转录共激活因子起作用[54]。HSP70在VGⅡ分离株中的扩增表明其可能是适应新环境的机制。进一步研究还发现,VGⅡ缺失了其他3个谱系中存在的约150个基因,比VGⅠ-Ⅲ-Ⅳ谱系中相应的基因丢失组合少3倍,进一步表明该谱系的基因组随着时间推移变得更加稳定。这些缺失基因富集的结构域包括PAZ、Piwi和DUF1785,所有这些都是RNA干扰机制的组成部分[55-56]。此外,VGⅡ中丢失的基因还包括参与蛋白质加工和降解的功能结构域,例如Alg14结构域(N-连接糖基化第二步所需)[57]、两个S9肽酶以及后期促进复合物亚基5。VGⅡ菌株还缺失了一半对核和线粒体基因组维持至关重要的PIF1样螺旋酶[58],这些可能在VGⅡ的不同管状线粒体形态中发挥作用。此外,过氧化物酶、细胞色素氧化酶c亚基VIb(COX6B)和铁蛋白铁结合区2也在VGⅡ分离株特异缺失,其可能参与了隐球菌的抗氧化应激反应[59]。
VGⅢ分离株约包含70个谱系特异基因,这个数目可能会随着VGⅢ测序菌株的增加而减少。目前发现,VGⅢ特异基因包括对基因组稳定性起重要作用的PIF1样解旋酶[58]和磷酸丙酮酸水合酶/烯醇酶[60],这是主要的高度保守的真菌过敏原[60]。此外,葡萄糖-甲醇-胆碱氧化还原酶,包括由多种真菌物种分泌的抗真菌蛋白,在VGⅢ中丢失[61]。含有亮氨酸拉链-EF-hand结构域跨膜蛋白1(leucine zipper/EF-hand- containing transmembrane protein 1,LETM1)也未在VGⅢ中预测到,尽管翻译的BLAST比对结果揭示了LETM1样序列的存在。
VGⅣ主要感染免疫功能低下的宿主,研究表明,VGⅣ具有最多的谱系特异基因,不过VGⅣ谱系菌株也特异缺失了一部分重要基因,如含有Ctr铜转运蛋白家族结构域的3个基因中的一个,这些基因已被证明可影响肺部感染期间的隐球菌存活和脑膜炎的发病[62]。VGⅣ缺失的基因还包括3种卤酸脱卤素酶的Pfam结构域,其在多种底物上催化碳或磷酰基转移反应[63]。
4.3单核苷酸多态性
目前,针对格特隐球菌菌株单核苷酸多态性(single nucleotide polymorphism,SNP)的分析主要集中于利用全基因组SNP进行格特隐球菌的进化研究,深入的细化和功能研究较少。2014年,Engelthaler等[22]首次对全球118株格特隐球菌菌株(主要为太平洋西北地区,115株VGⅡ谱系菌株以及VGⅠ、VGⅢ和VGⅣ谱系各一株)进行了全基因组SNP比较分析,在所有基因组中共鉴定出1 282 876个SNP,544 881个简约SNP(区分不止一个菌株);在115个VGⅡ基因组中,发现310 969个SNP,其中221 248个简约SNP;对太平洋西北地区爆发株不同亚型的分析显示,VGⅡa、VGⅡb和VGⅡc分别仅包含717、246和528个SNP。进一步研究还表明,具有更高毒力的VGⅡa群体来自毒力较弱的谱系,该谱系在MSH2直系同源基因中存在突变,但随后在VGⅡa株中恢复,表现出瞬时突变体表型,可能有助于其适应环境并增加毒力[23]。
5 结语
格特隐球菌是近年来受到关注的人类重要的病原性真菌之一,随着基因组测序技术的不断发展和成熟,格特隐球菌的基因组学研究逐渐引起学术界关注。基于新一代高通量测序技术的全基因组测序,不仅可更好地了解格特隐球菌基因组特征,为基因功能研究提供指导;同时在格特隐球菌的致病性和防治方面,还可进行致病相关基因鉴定,促进疫苗以及新型抗生素的开发。格特隐球菌的基因组学研究必将在隐球菌病的防治工作中发挥重要作用。
参考文献
[1]Kwon-Chung KJ, Varma A. Do major species concepts support one, two or more species within Cryptococcus neoformans?[J]. FEMS Yeast Res, 2006, 6: 574-587.
[2]Chen SCA, Meyer W, Sorrell TC. Cryptococcus gattii Infections[J]. Clin Microbiol Rev, 2014, 27: 980-1024.
[3]Chen CH, Wang SH, Chen WL, et al. Ocular complications caused by Cryptococcus gattii AFLP4/VGI meningitis in an immunocompetent host[J]. Rev Inst Med Trop Sao Paulo, 2016, 58: 85.
[4]Meyer W. Cryptococcus gattii in the age of whole-genome sequencing[J]. Mbio, 2015, 6:e01761-15.
[5]Bovers M, Hagen F, Kuramae EE, et al. Six monophyletic lineages identified within Cryptococcus neoformans and Cryptococcus gattii by multi-locus sequence typing[J]. Fungal Genet Biol, 2008, 45: 400-421.
[6]Kidd SE, Hagen F, Tscharke RL, et al. A rare genotype of Cryptococcus gattii caused the cryptococcosis outbreak on Vancouver Island (British Columbia, Canada)[J]. Proc Natl Acad Sci U S A, 2004, 101: 17258-17263.
[7]Meyer W, Castaneda A, Jackson S, et al. Molecular typing of IberoAmerican Cryptococcus neoformans isolates[J]. Emerg Infect Dis, 2003, 9: 189-195.
[8]Trilles L, Lazera M, Wanke B, et al.Genetic characterization of environmental isolates of the Cryptococcus neoformans species complex from Brazil[J]. Med Mycol, 2003, 41: 383-390.
[9]Trilles L, Lazera Mdos S, Wanke B, et al. Regional pattern of the molecular types of Cryptococcus neoformans and Cryptococcus gattii in Brazil[J]. Mem I Oswaldo Cruz, 2008, 103: 455-462.
[10]Harris JR, Lockhart SR, Debess E, et al. Cryptococcus gattii in the United States: clinical aspects of infection with an emerging pathogen[J]. Clinl Infect Dis,2011, 53: 1188-1195.
[11]窦红涛, 万喆, 杨启文,等. 格特隐球菌在河北地区引起 1 例脑膜炎的临床与实验研究[J]. 中国真菌学杂志, 2015, 10: 11-15.
[12]金亮, 沈定霞. 格特隐球菌感染的临床特征与实验室研究进展[J]. 检验医学, 2017, 32: 1065-1069.
[13]葛瑛,张凯宇,马小军,等. 隐球菌脑膜炎62例临床分析[J]. 协和医学杂志, 2018, 9:431-436.
[14]Velez N, Escandon P. Report on novel environmental niches for Cryptococcus neoformans and Cryptococcus gattii in Colombia: Tabebuia guayacan and Roystonea regia[J]. Med Mycol, 2017, 55: 794-797.
[15]DSouza CA, Kronstad JW, Taylor G, et al. Genome variation in Cryptococcus gattii, an emerging pathogen of immunocompetent hosts[J]. Mbio, 2011, 2: e00342-10.
[16]Bovers M, Hagen F, Kuramae EE, et al. Promiscuous mitochondria in Cryptococcus gattii[J]. FEMS Yeast Res, 2009, 9: 489-503.
[17]Ngamskulrungroj P, Gilgado F, Faganello J, et al. Genetic diversity of the Cryptococcus species complex suggests that Cryptococcus gattii deserves to have varieties[J]. PLoS One, 2009, 4: e5862.
[18]Xu J, Vilgalys R, Mitchell TG. Multiple gene genealogies reveal recent dispersion and hybridization in the human pathogenic fungus Cryptococcus neoformans[J]. Mol Ecol, 2000, 9: 1471-1481.
[19]Gonzalez GM, Casillas-Vega N, Garza-Gonzalez E, et al. Molecular typing of clinical isolates of Cryptococcus neoformans/Cryptococcus gattii species complex from Northeast Mexico[J]. Folia Microbiol, 2016, 61: 51-56.
[20]Gillece JD, Schupp JM, Balajee SA, et al. Whole genome sequence analysis of Cryptococcus gattii from the Pacific Northwest reveals unexpected diversity[J]. PLoS One, 2011, 6: e28550.
[21]Hagen F, Ceresini PC, Polacheck I, et al. Ancient dispersal of the human fungal pathogen Cryptococcus gattii from the Amazon rainforest[J]. PLoS One, 2013, 8: e71148.
[22]Engelthaler DM, Hicks ND, Gillece JD, et al. Cryptococcus gattii in North American Pacific Northwest: Whole-popula-tion genome analysis provides insights into species evolution and dispersal[J]. Mbio, 2014, 5: e01464-14.
[23]Billmyre RB, Croll D, Li WJ, et al. Highly recombinant VGII Cryptococcus gattii population develops clonal outbreak clusters through both sexual macroevolution and asexual microevolution[J]. Mbio, 2014, 5: e01494-14.
[24]Byrnes EJ, Bildfell RJ, Frank SA, et al. Molecular evidence that the range of the Vancouver Island out-break of Cryptococcus gattii infection has expanded into the Pacific Northwest in the United States[J]. J Infect Dis, 2009, 199:1081-1086.
[25]Hagen F, Colom MF, Swinne D, et al. Autochthonous and dormant Cryptococcus gattii infections in Europe[J]. Emerg Infect Dis, 2012, 18: 1618-1624.
[26]Steele KT, Thakur R, Nthobatsang R, et al. In-hospital mortality of HIV-infected cryptococcal meningitis patients with C. gattii and C. neoformans infection in Gaborone, Botswana[J]. Med Mycol, 2010, 48: 1112-1115.
[27]Carriconde F, Gilgado F, Arthur I, et al. Clonality and alpha-a recombination in the Australian Cryptococcus gattii VGII population—an emerging outbreak in Australia[J]. PLoS One, 2011, 6: e16936.
[28]Kronstad JW, Attarian R, Cadieux B, et al. Expanding fungal pathogenesis: Cryptococcus breaks out of the opportunistic box[J]. Nat Rev Microbiol, 2011, 9: 193-203.
[29]Ma H, May RC. Virulence in Cryptococcus species[J]. Adv Appl Microbiol, 2009, 67: 131-190.
[30]Kozel TR, Gotschlich EC. The capsule of Cryptococcus neoformans passively inhibits phagocytosis of the yeast by macrophages[J]. J Immunol, 1982, 129: 1675-1680.
[31]Tucker SC, Casadevall A. Replication of Cryptococcus neoformans in macrophages is accompanied by phagosomal permeabilization and accumulation of vesicles containing polysaccharide in the cytoplasm[J]. Proc Natl Acad Sci U S A, 2002, 99: 3165-3170.
[32]Feldmesser M, Kress Y, Novikoff P, et al.Cryptococcus neoformans is a facultative intracellular pathogen in murine pulmonary infection[J]. Infect Immun, 2000, 68: 4225-4237.
[33]Casadevall A, Rosas AL, Nosanchuk JD. Melanin and virulence in Cryptococcus neoformans[J]. Curr Opin Microbiol, 2000, 3: 354-358.
[34]Williamson PR. Laccase and melanin in the pathogenesis of Cryptococcus neoformans[J]. Frontiers Biosci, 1997, 2: e99-e107.
[35]van Duin D, Casadevall A, Nosanchuk JD. Melanization of Cryptococcus neoformans and histoplasma capsulatum reduces their susceptibilities to amphotericin B and caspofungin[J]. Antimicrob Agents Ch, 2002, 46: 3394-3400.
[36]Perfect JR. Cryptococcus neoformans: A sugar-coated killer with designer genes[J]. Fems Immunol Med Mic, 2005, 45: 395-404.
[37]Ghannoum MA. Potential role of phospholipases in virulence and fungal pathogenesis[J]. Clin Microbiol Rev, 2000, 13: 122-143.
[38]Cox GM, McDade HC, Chen SCA, et al. Extracellular phospholipase activity is a virulence factor for Cryptococcus neoformans[J]. Mol Microbiol, 2001, 39: 166-175.
[39]Steenbergen JN, Casadevall A. The origin and maintenance of virulence for the human pathogenic fungus Cryptococcus neoformans[J]. Microbes Infect, 2003, 5: 667-675.
[40]Ganendren R, Carter E, Sorrell T, et al.Phospholipase B activity enhances adhesion of Cryptococcus neoformans to a human lung epithelial cell line[J]. Microbes Infect, 2006, 8: 1006-1015.
[41]Santangelo R, Zoellner H, Sorrell T, et al. Role of extracellular phospholipases and mononuclear phagocytes in dissemination of cryptococcosis in a murine model[J]. Infect Immun, 2004, 72: 2229-2239.
[42]Cox GM, Mukherjee J, Cole GT, et al.Urease as a virulence factor in experimental cryptococcosis[J]. Infect Immun, 2000, 68: 443-448.
[43]Kwonchung KJ, Bennett JE. Distribution of alpha and a mating types of Cryptococcus neoformans among natural and clinical isolates[J]. Am J Epidemiol, 1978, 108: 337-340.
[44]Kwon-Chung KJ, Edman JC, Wickes BL. Genetic association of mating types and virulence in Cryptococcus neoformans[J]. Infect Immun, 1992, 60: 602-605.
[45]Karos M, Chang YC, McClelland CM, et al. Mapping of the Cryptococcus neoformans MAT alpha locus: Presence of mating type-specific mitogen-activated protein kinase cascade homologs[J]. J Bacteriol, 2000, 182: 6222-6227.
[46]Fraser JA, Heitman J. Evolution of fungal sex chromosomes[J]. Mol Microbiol, 2004, 51: 299-306.
[47]Farrer RA, Desjardins CA, Sakthikumar S, et al. Genome evolution and innovation across the four major lineages of Cryptococcus gattii[J]. Mbio, 2015, 6: e00868-15.
[48]Janbon G, Ormerod KL, Paulet D, et al. Analysis of the genome and transcriptome of Cryptococcus neoformans var. grubii reveals complex RNA expression and microevolution leading to virulence attenuation[J]. PLoS Genet, 2014, 10: e1004261.
[49]Hu G, Wang J, Choi J, et al. Variation in chromosome copy number influences the virulence of Cryptococcus neoformans and occurs in isolates from AIDS patients[J]. BMC Genomics, 2011, 12: 526.
[50]Hudson RR. Genetic data analysis: Methods for discrete population genetic data[J]. Science, 1990, 250: 575.
[51]Saikia S, Oliveira D, Hu G, et al.Role of ferric reductases in iron acquisition and virulence in the fungal pathogen Cryptococcus neoformans[J]. Infect Immun, 2014, 82: 839-850.
[52]Aravind L, Koonin EV. The HORMA domain: a common structural denominator in mitotic checkpoints, chromosome synapsis and DNA repair[J]. Trends Biochem Sci, 1998, 23: 284-286.
[53]van Dijk TB, Gillemans N, Stein C, et al. Friend of Prmt1, a novel chromatin target of protein arginine methyltrans-ferases[J]. Mol cell biol, 2010, 30: 260-272.
[54]Zhang S, Hacham M, Panepinto J, et al. The Hsp70 member, Ssa1, acts as a DNA-binding transcriptional co-activ-ator of laccase in Cryptococcus neoformans[J]. Mol Microbiol, 2006, 62: 1090-1101.
[55]Janbon G, Maeng S, Yang DH, et al. Characterizing the role of RNA silencing components in Cryptococcus neoformans[J]. Fungal Genet Biol, 2010, 47: 1070-1080.
[56]Wang X, Hsueh YP, Li W, et al.Sex-induced silencing defends the genome of Cryptococcus neoformans via RNAi[J]. Gene Dev, 2010, 24: 2566-2582.
[57]Gao XD, Tachikawa H, Sato T, et al. Alg14 recruits Alg13 to the cytoplasmic face of the endoplasmic reticulum to form a novel bipartite UDP-N-acetylglucosamine transferase required for the second step of N-linked glycosylation[J]. J Biol Chem, 2005, 280: 36254-36262.
[58]Bochman ML, Sabouri N, Zakian VA. Unwinding the functions of the Pif1 family helicases[J]. DNA Repair, 2010, 9: 237-249.
[59]Giles SS, Perfect JR, Cox GM. Cytochrome c peroxidase contributes to the antioxidant defense of Cryptococcus neoformans[J]. Fungal Genet Biol, 2005, 42: 20-29.
[60]Breitenbach M, Simon B, Probst G, et al. Enolases are highly conserved fungal allergens[J]. Int Arch Allergy Imm, 1997, 113: 114-117.
[61]Kawabe M, Okabe Onokubo A, Arimoto Y, et al. GMC oxidoreductase, a highly expressed protein in a potent biocontrol agent Fusarium oxysporum Cong:1-2, is dispensable for biocontrol activity[J]. J Gen Appl Microbiol, 2011, 57: 207-217.
[62]Sun TS, Ju X, Gao HL, et al. Reciprocal functions of Cryptococcus neoformans copper homeostasis machinery during pulmonary infection and meningoencephalitis[J]. Nat Commun, 2014, 5: 5550.
[63]Burroughs AM, Allen KN, Dunaway-Mariano D, et al.Evolutionary genomics of the HAD superfamily: understanding the structural adaptations and catalytic diversity in a superfamily of phosphoesterases and allied enzymes[J]. J Mol Biol, 2006, 361: 1003-1034.
作者:贾鑫淼1,3,肖盟2,3,孙天舒1,3,徐英春2,3
单位:北京协和医院 1中心实验室 2检验科 3侵袭性真菌病机制研究与精准诊断北京市重点实验
通信作者:徐英春
文章来源:协和医学杂志,2019,10(5):505 - 513.
第一作者简介
贾鑫淼
博士,生物信息学,北京协和医院-中心实验室-统计与生信平台。
北京协和医院-中心实验室-统计与生信平台基于循证医学、医学统计学、临床流行病学、生物信息学理论与前沿技术,在院内外开展方法学咨询服务与大数据分析技术支持。从疾病的病因、诊断、治疗、预后多角度开展临床研究,为疾病预防、早期干预、新药和新治疗方法的临床应用提供科学的证据支持,促进转化医学发展与科研成果转化。
欢迎有兴趣的同道与我们联系,联系方式:pumchstat@126.com。
通信作者简介
徐英春
研究员、教授、博士研究生导师,北京协和医院检验科主任,临床生物标本管理中心主任,临床检验诊断学系主任。
主要学术兼职:
欧洲临床抗菌药物敏感实验委员会(EUCAST)华人抗菌药物敏感实验(ChiCAS)分委会主任委员,欧洲临床微生物感染疾病学会(ESCMID)合作实验室主任,世界华人检验舆病理医师协会副会长,国家卫生计生委抗菌药物临床应用与细菌耐药评价专家委员委员兼办公室主任,国家卫计委合理用药专家委员会抗菌药物组副组长,国家卫计委全国细菌耐药监测网质量管理中心负责人,中国医师协会检验医师分会常委。
版权声明:
协和医学杂志倡导尊重和保护知识产权。欢迎转载、引用,但需取得本平台授权。如您对文章内容版权存疑,请发送邮件medj@pumch.cn,我们会与您及时沟通处理。本站内容及图片仅供参考、学习使用,不为盈利且不作为诊断、医疗根据。




 扫码下载APP
扫码下载APP

 科普中国APP
科普中国APP
 科普中国
科普中国
 科普中国
科普中国