 你发现了没有,在科幻电影中,平民出身的“超级英雄”都有一个共同点:他们的超能力往往不是靠勤学苦练练出来的,而都是靠修改基因改出来的!
你发现了没有,在科幻电影中,平民出身的“超级英雄”都有一个共同点:他们的超能力往往不是靠勤学苦练练出来的,而都是靠修改基因改出来的! 虽然超能力在现实中并不存在,但是修改人类的基因早已成为了现实。今天咱们就来说说,新闻里常说的基因编辑是怎么回事?编辑基因会有啥好处?(一)先说说基因是啥。基因说白了,就是制造生物体的图纸。生物体的大部分细胞里都有这样一整套的基因图纸。蜘蛛侠之所以能吐丝,能飞檐走壁,靠的就是基因图纸的指导。
虽然超能力在现实中并不存在,但是修改人类的基因早已成为了现实。今天咱们就来说说,新闻里常说的基因编辑是怎么回事?编辑基因会有啥好处?(一)先说说基因是啥。基因说白了,就是制造生物体的图纸。生物体的大部分细胞里都有这样一整套的基因图纸。蜘蛛侠之所以能吐丝,能飞檐走壁,靠的就是基因图纸的指导。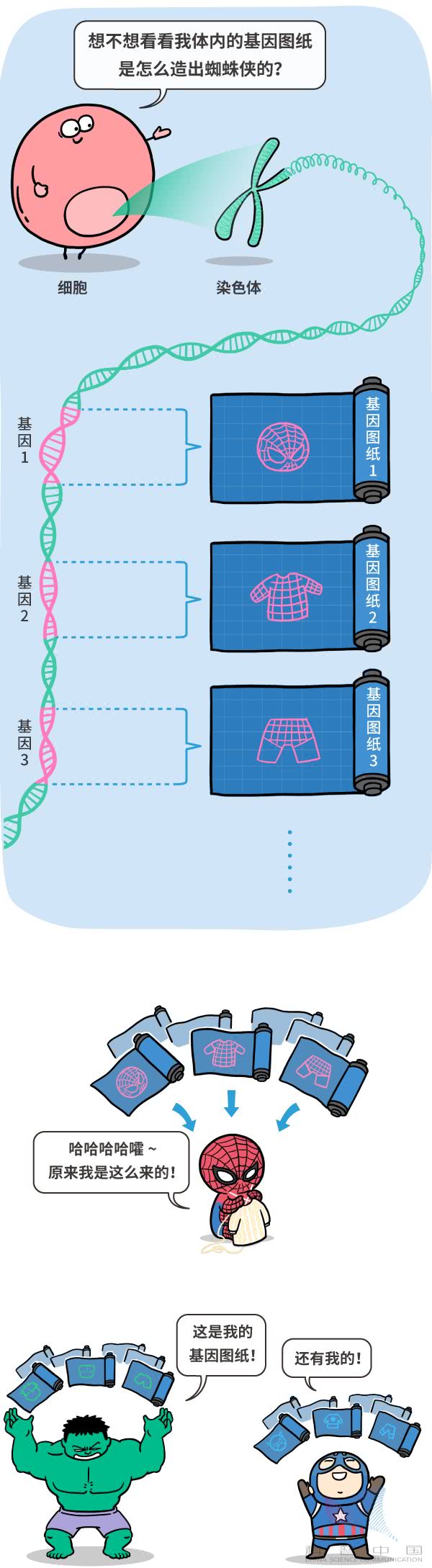 只不过,基因这种图纸,不是直接把生物特征画成了图,而是将它加密成了一串密码。这串密码也不是用普通人能看得懂的符号写的,而是由四种特殊的化学分子排列成的。这四种化学分子分别记作A、T、C、G [注:1]。
只不过,基因这种图纸,不是直接把生物特征画成了图,而是将它加密成了一串密码。这串密码也不是用普通人能看得懂的符号写的,而是由四种特殊的化学分子排列成的。这四种化学分子分别记作A、T、C、G [注:1]。 在细胞中,这串由化学分子排列形成的密码长链就是我们平常说的
在细胞中,这串由化学分子排列形成的密码长链就是我们平常说的 所以,基因编辑,就是像我们编辑手机中的文字一样,编辑 DNA 分子中某几个特定的化学分子!那么,基因编辑跟我们普通人又有什么关系呢?(二)人会有缺陷,计算机程序会有缺陷。
所以,基因编辑,就是像我们编辑手机中的文字一样,编辑 DNA 分子中某几个特定的化学分子!那么,基因编辑跟我们普通人又有什么关系呢?(二)人会有缺陷,计算机程序会有缺陷。 同样的道理,生物细胞里存放的基因图纸也会有缺陷。
同样的道理,生物细胞里存放的基因图纸也会有缺陷。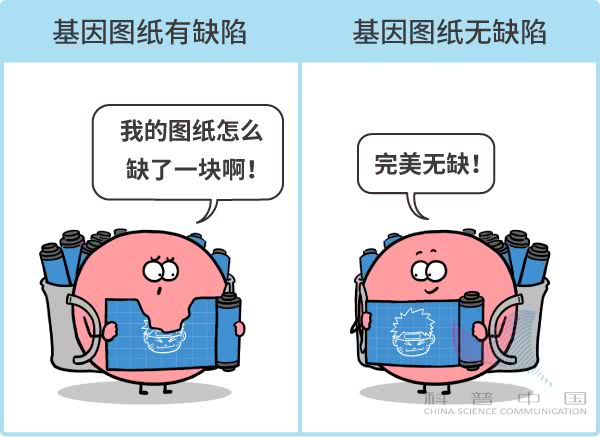 有时候,基因缺陷的后果很严重。因为图纸出了问题,造出的零件就可能出问题。零件出了问题,使用零件的机器就可能出问题。最严重的时候,整个“生命工厂”都会因此产生重大事故。
有时候,基因缺陷的后果很严重。因为图纸出了问题,造出的零件就可能出问题。零件出了问题,使用零件的机器就可能出问题。最严重的时候,整个“生命工厂”都会因此产生重大事故。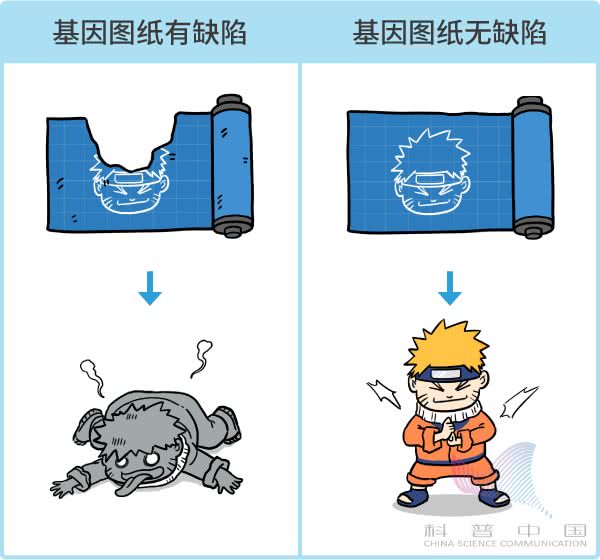 实际上,许多打针吃药做手术都治不好的病,都是跟基因缺陷有关。比如,有一种病叫作联合免疫缺陷。得了这种病的小孩,免疫系统几乎完全失灵,通常只能住在与世隔绝的气泡舱里。这种病的一种较常见的原因是 ADA 基因存在缺陷。
实际上,许多打针吃药做手术都治不好的病,都是跟基因缺陷有关。比如,有一种病叫作联合免疫缺陷。得了这种病的小孩,免疫系统几乎完全失灵,通常只能住在与世隔绝的气泡舱里。这种病的一种较常见的原因是 ADA 基因存在缺陷。 除此之外,癌症、血友病、白化病等等,甚至某些高血压和糖尿病,都跟基因缺陷有关。所以,就算我们不想制造超级英雄,为了身体健康,我们也得了解一点基因编辑的知识。(三)要想搞清楚基因该怎么编辑,就得先分清楚基因存在哪类缺陷。粗略地说,基因缺陷可以分为两类。一类叫作“该有的没有”,另一类叫作“不该有的乱有”。1.该有的没有假如基因缺陷导致某个基因造出的零件不好用,或者干脆啥也造不出来,就属于该有的没有。此时,科学家就会往细胞里添一个好用的基因,也就是“缺啥补啥”[1]。这种任务通常由某种改造过的病毒来完成。说白了,就是让病毒把好用的基因塞到细胞里。
除此之外,癌症、血友病、白化病等等,甚至某些高血压和糖尿病,都跟基因缺陷有关。所以,就算我们不想制造超级英雄,为了身体健康,我们也得了解一点基因编辑的知识。(三)要想搞清楚基因该怎么编辑,就得先分清楚基因存在哪类缺陷。粗略地说,基因缺陷可以分为两类。一类叫作“该有的没有”,另一类叫作“不该有的乱有”。1.该有的没有假如基因缺陷导致某个基因造出的零件不好用,或者干脆啥也造不出来,就属于该有的没有。此时,科学家就会往细胞里添一个好用的基因,也就是“缺啥补啥”[1]。这种任务通常由某种改造过的病毒来完成。说白了,就是让病毒把好用的基因塞到细胞里。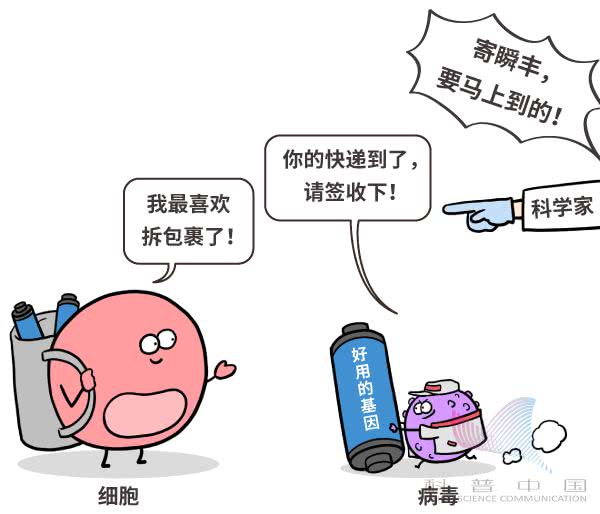 比如,1990 年,美国国立卫生院临床中心就是用添基因的办法(即添加正常的 ADA 基因),治好了一个患有联合免疫缺陷的小女孩。于是,这个小女孩再也不用在气泡舱里生活了。
比如,1990 年,美国国立卫生院临床中心就是用添基因的办法(即添加正常的 ADA 基因),治好了一个患有联合免疫缺陷的小女孩。于是,这个小女孩再也不用在气泡舱里生活了。 话说回来,往细胞里添基因虽然也属于基因编辑,但这种方法有点儿不够精准。因为用这种方法添加的基因,会在 DNA 密码链中随便找地方落脚,有点儿不受控制。相比之下,下面这种情况才是手术刀级别的“基因编辑”。2.不该有的乱有假如基因缺陷的后果不是零件不好用,而是零件功能错乱,导致不该有的乱有,那么,添基因的办法就不管用了。
话说回来,往细胞里添基因虽然也属于基因编辑,但这种方法有点儿不够精准。因为用这种方法添加的基因,会在 DNA 密码链中随便找地方落脚,有点儿不受控制。相比之下,下面这种情况才是手术刀级别的“基因编辑”。2.不该有的乱有假如基因缺陷的后果不是零件不好用,而是零件功能错乱,导致不该有的乱有,那么,添基因的办法就不管用了。 这个时候,科学家必须把基因的缺陷修改掉,也就是像编辑图纸中的错别字/错线段一样,直接在 DNA 密码链特定的位置上插入、删除、替换其中的某几个密码字符(即“化学分子”),才能彻底解决这个问题。这就是手术刀级别的基因编辑。
这个时候,科学家必须把基因的缺陷修改掉,也就是像编辑图纸中的错别字/错线段一样,直接在 DNA 密码链特定的位置上插入、删除、替换其中的某几个密码字符(即“化学分子”),才能彻底解决这个问题。这就是手术刀级别的基因编辑。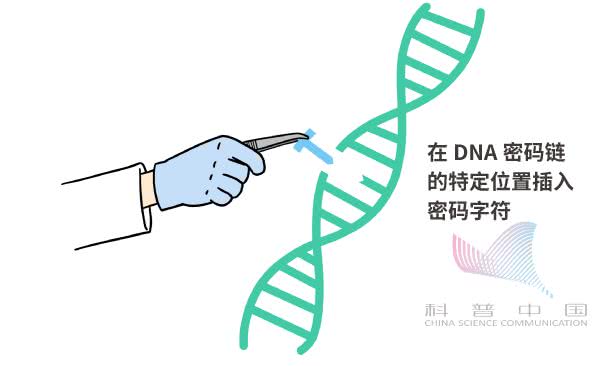 比如,有一种叫作 HTT 的基因包含 9000 多个密码字符,其中的字符难免会出错(注:多出来好几处内容为“ CAG ”的密码序列)。如果“错别字”数量超过一定阈值,人类和小鼠就会得一种奇怪的遗传病,叫作亨廷顿舞蹈症。
比如,有一种叫作 HTT 的基因包含 9000 多个密码字符,其中的字符难免会出错(注:多出来好几处内容为“ CAG ”的密码序列)。如果“错别字”数量超过一定阈值,人类和小鼠就会得一种奇怪的遗传病,叫作亨廷顿舞蹈症。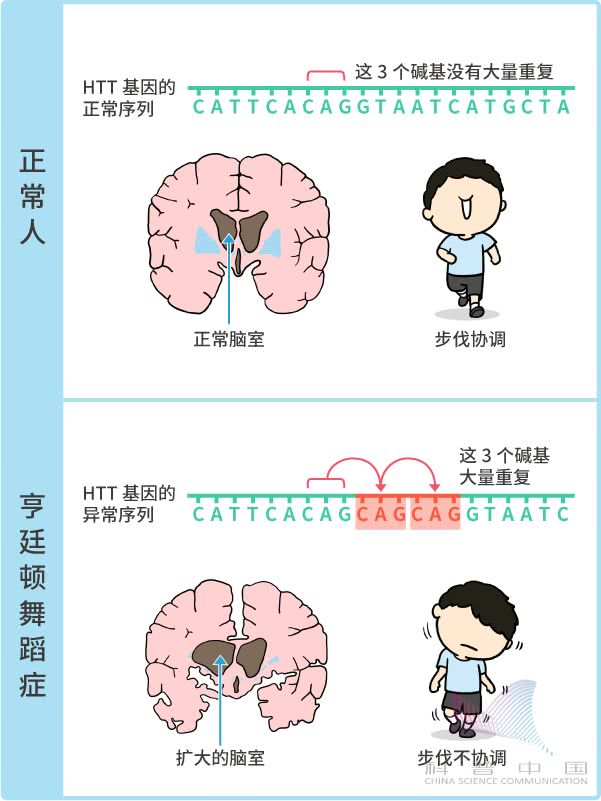 科学家发现,利用基因编辑技术,把 DNA 密码链中的“错别字”编辑掉,小鼠的亨廷顿舞蹈症就能治好[2]!(四)在人类的 DNA 中,密码字符的数量高达几十亿个
科学家发现,利用基因编辑技术,把 DNA 密码链中的“错别字”编辑掉,小鼠的亨廷顿舞蹈症就能治好[2]!(四)在人类的 DNA 中,密码字符的数量高达几十亿个 怎样才能改正其中的“错别字”呢?可以设想,不管用什么办法,这个过程一定会分成三个步骤:
怎样才能改正其中的“错别字”呢?可以设想,不管用什么办法,这个过程一定会分成三个步骤: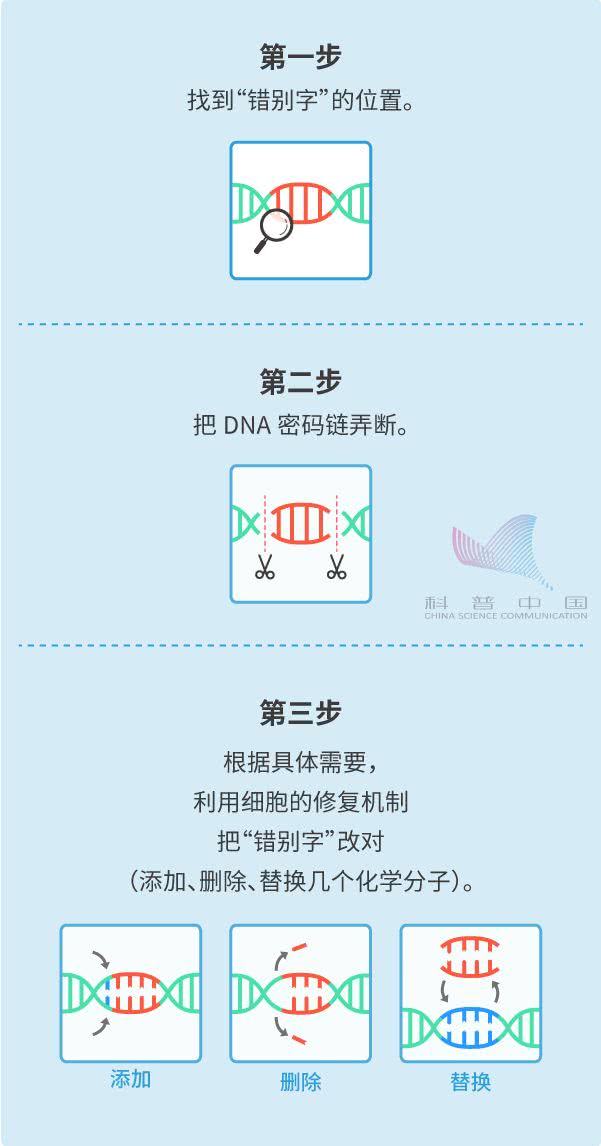 所以,科学家要想实现基因编辑,就需要有一套好用的工具,配合细胞修复机制,共同完成这几个步骤。那么,世界上真的存在这么好用的工具吗?(五)这样的工具真的存在,而且,它们往往存在于自然界的生物细胞中。例如,最近格外出名的基因编辑工具CRISPR/Cas 系统,就是在细菌体内发现的。CRISPR/Cas 原本是细菌的免疫系统,最擅长剪断特定的 DNA 。经过科学家改造后,它就可以帮助我们编辑指定基因了。
所以,科学家要想实现基因编辑,就需要有一套好用的工具,配合细胞修复机制,共同完成这几个步骤。那么,世界上真的存在这么好用的工具吗?(五)这样的工具真的存在,而且,它们往往存在于自然界的生物细胞中。例如,最近格外出名的基因编辑工具CRISPR/Cas 系统,就是在细菌体内发现的。CRISPR/Cas 原本是细菌的免疫系统,最擅长剪断特定的 DNA 。经过科学家改造后,它就可以帮助我们编辑指定基因了。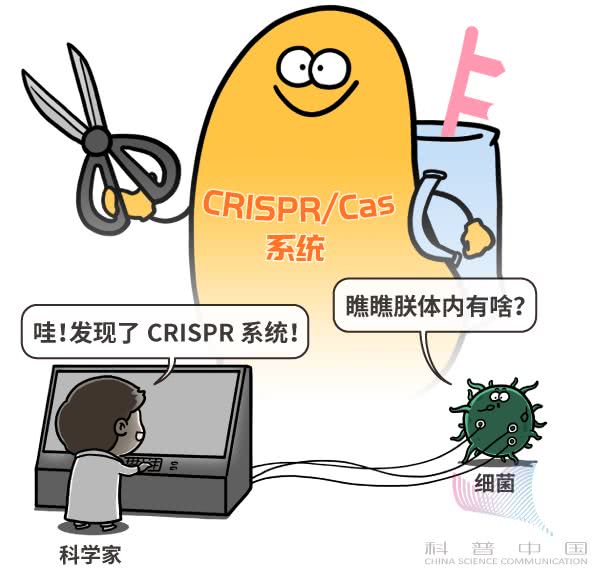 比方说,在这套工具中,专门有一串化学分子(向导RNA)可以在 DNA 密码链中识别“目标错别字”。一旦它找到了“目标错别字”,就会像拉链一样,跟一条 DNA 密码链紧紧地扣在一起。这就是第一步。
比方说,在这套工具中,专门有一串化学分子(向导RNA)可以在 DNA 密码链中识别“目标错别字”。一旦它找到了“目标错别字”,就会像拉链一样,跟一条 DNA 密码链紧紧地扣在一起。这就是第一步。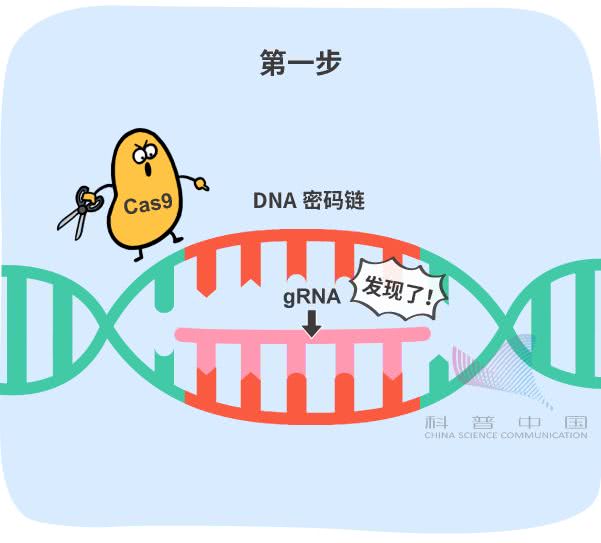 此时,有一种和它组合在一起的蛋白质(Cas9)就会对准DNA密码链的这个位置,“咔嚓”剪一刀。这就是第二步。
此时,有一种和它组合在一起的蛋白质(Cas9)就会对准DNA密码链的这个位置,“咔嚓”剪一刀。这就是第二步。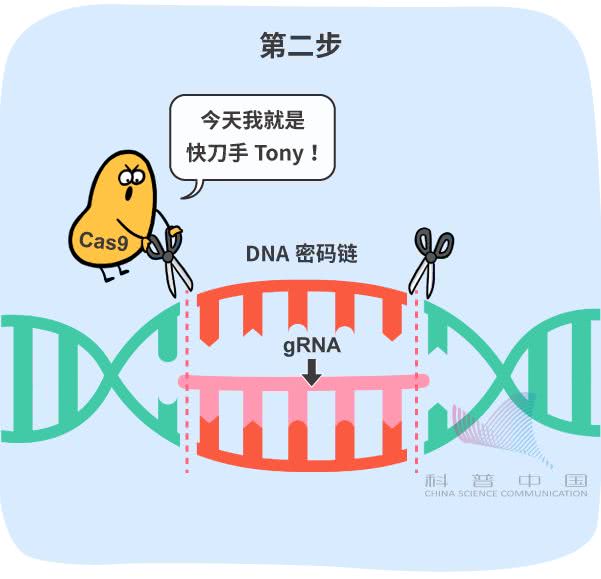 最后,细胞的修复机制会发挥作用,按照科学家指定的“内容模板”,把“错别字”改对,再把 DNA 密码链接好。这就是第三步。
最后,细胞的修复机制会发挥作用,按照科学家指定的“内容模板”,把“错别字”改对,再把 DNA 密码链接好。这就是第三步。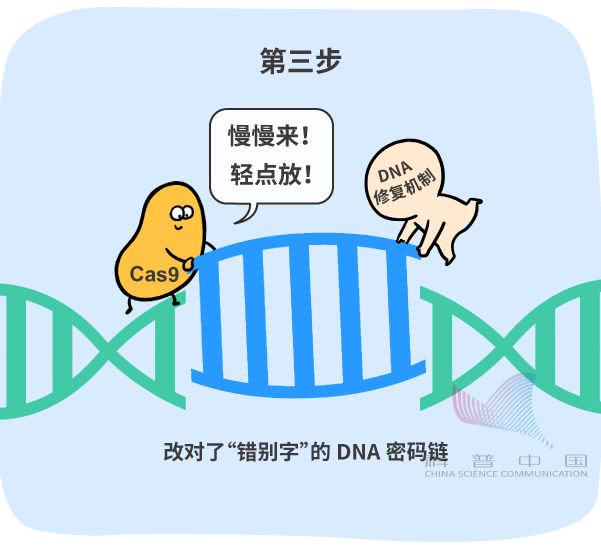 咱们在上文说的编辑小鼠 HTT 基因,治疗小鼠亨廷顿舞蹈症的实验,用的就是这套工具(CRISPR/Cas 系统),是不是很厉害?
咱们在上文说的编辑小鼠 HTT 基因,治疗小鼠亨廷顿舞蹈症的实验,用的就是这套工具(CRISPR/Cas 系统),是不是很厉害? 可惜,目前这种工具还不够成熟,暂时还没有用在人体的基因编辑中。(六)虽然 CRISPR/Cas 系统现在风头正劲,但它目前还存在两个不足:一是脱靶效应,二是缺乏默契。第一个不足是脱靶效应。也就是说,在它识别错别字的时候,可能会误伤某段正常的密码链(比如,一段密码链的内容跟“目标错别字”很像,但不完全一样时)。
可惜,目前这种工具还不够成熟,暂时还没有用在人体的基因编辑中。(六)虽然 CRISPR/Cas 系统现在风头正劲,但它目前还存在两个不足:一是脱靶效应,二是缺乏默契。第一个不足是脱靶效应。也就是说,在它识别错别字的时候,可能会误伤某段正常的密码链(比如,一段密码链的内容跟“目标错别字”很像,但不完全一样时)。 这个道理很简单。CRISPR/Cas 系统本来就是细菌的“锄奸队”,它的任务是铲除病毒植入的病毒基因。由于病毒的基因会不断变异,所以它得有一定的灵活度,不能总是要求100%的匹配。
这个道理很简单。CRISPR/Cas 系统本来就是细菌的“锄奸队”,它的任务是铲除病毒植入的病毒基因。由于病毒的基因会不断变异,所以它得有一定的灵活度,不能总是要求100%的匹配。 而且,由于人的某些基因是成对地出现在一对染色体上的。当一对基因都存在缺陷时,编辑基因的任务量就会翻倍,难度也会随之增加。第二个不足是缺乏默契。CRISPR/Cas 系统再怎么经过改造,它也是从细菌来的。细菌没有细胞核,跟人这样的有细胞核的生物非常不同。CRISPR/Cas 系统跟人体细胞修复机制打配合的时候,就好比让C罗跟篮球队员打配合,肯定会缺乏默契。
而且,由于人的某些基因是成对地出现在一对染色体上的。当一对基因都存在缺陷时,编辑基因的任务量就会翻倍,难度也会随之增加。第二个不足是缺乏默契。CRISPR/Cas 系统再怎么经过改造,它也是从细菌来的。细菌没有细胞核,跟人这样的有细胞核的生物非常不同。CRISPR/Cas 系统跟人体细胞修复机制打配合的时候,就好比让C罗跟篮球队员打配合,肯定会缺乏默契。 所以,某些 CRISPR/Cas 编辑工具把 DNA 剪断以后,细胞会胡乱补一通,结果导致“错别字”不但没改对,反而成了“错上加错”。(七)当然,正是因为技术还不够成熟,科学家才要继续研究它。截至 2017 年底,科学家正在进行或已完成的基因治疗临床试验高达 2400 多项。这些试验有的想要治疗癌症(64.6%),有的想要治疗单个基因引起的遗传病(10.5%),有的想要治疗传染病和心血管疾病等跟多个基因有关的疾病(7.4%)。其中一些试验,就需要用到基因编辑技术。
所以,某些 CRISPR/Cas 编辑工具把 DNA 剪断以后,细胞会胡乱补一通,结果导致“错别字”不但没改对,反而成了“错上加错”。(七)当然,正是因为技术还不够成熟,科学家才要继续研究它。截至 2017 年底,科学家正在进行或已完成的基因治疗临床试验高达 2400 多项。这些试验有的想要治疗癌症(64.6%),有的想要治疗单个基因引起的遗传病(10.5%),有的想要治疗传染病和心血管疾病等跟多个基因有关的疾病(7.4%)。其中一些试验,就需要用到基因编辑技术。 虽然现在基因编辑技术在科学、法律和伦理等层面仍然面临着许多挑战,但种种结果显示,用它治疗疾病的潜力非常大。要想改善人们的健康,将基因编辑真正地用在实处,我们不仅需要科学家的努力,还需要全社会一起努力。作者 | Sheldon 科学漫画工作室审稿 | MarvinP 加州大学洛杉矶分校研究员责编 | 高佩雯文章由腾讯科普“科普中国头条创作与推送项目”团队推出转载请注明来自科普中国注释:1. 在生物学中,A、T、G、C分别代表腺嘌呤(A)、胸腺嘧啶(T)、鸟嘌呤(G)、胞嘧啶(C)。生物学家常常把它们统称为碱基。在DNA分子中,碱基是成对出现的。其中,A和T组成一对,G和C组成一对。所以,一个DNA分子,就是由配对碱基组成的两行密码序列。并且,这两行密码序列保存的信息内容是完全等价的。当一个细胞分裂成两个细胞的时候,DNA分子就会以这两行密码序列为蓝本,进行自我复制,复制出两个一模一样的DNA分子,然后把它们分配给两个细胞。2. 一个DNA分子上包含大量的碱基序列,也就是包含大量的密码序列。其中一些密码序列读取以后,什么零件也造不出来,并不直接影响生物体的形成和发育,叫作非编码DNA。另一些密码序列会被细胞当作零件的图纸来读取,这些序列就叫作基因。细胞读取了基因图纸以后,就会参照图纸的说明,制造出相应的零件。其中每一种零件,就对应一种拥有特定功能的蛋白质。这些蛋白质有的变成了生物体的一部分,有的负责调控生物体的生长和发育。一个生物体的全部遗传信息,就叫作这种生物体的基因组。3. 3月1日,中科院神经所的杨辉团队在《科学》杂志上发表了一篇论文,用一种新技术(Genome-wide Off-target analysis by Two-cell embryo Injection)检测了几种基因编辑工具存在的缺陷。他们发现,CRISPR/Cas系统本身产生“错上加错”的情况并没有想象中的严重,但科学家改造它时给它额外添加的“剪辑编辑器”(BE3.0)会产生非常多的“错上加错”的结果。4. 本漫画已对基因编辑的过程做了大幅简化。实际过程跟科学家对“CRISPR/cas系统”的改造手段有关,也跟编辑基因时具体的需求有关。参考文献:1.王立铭,《上帝的手术刀》,浙江人民出版社。2.M. Eisenstein, CRISPR takes on Huntington’s disease, Nature 557, S42-S43 (2018).3.Dunbar CE et al., Gene therapy comes of age, Science, Vol. 359, Issue 6372, eaan4672.4.何东明, 中国学者在Science发表两篇论文,同时揭示碱基编辑器脱靶奥秘. http://www.zhishifenzi.com/news/biology/5322.html5.E. Zuo et al., Cytosine base editor generates substantial off-target single nucleotide variants in mouse embryos, Science 10.1126/science.aav9973 (2019).6.S. Jin et al., Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice, Science 10.1126/science.aaw7166 (2019).
虽然现在基因编辑技术在科学、法律和伦理等层面仍然面临着许多挑战,但种种结果显示,用它治疗疾病的潜力非常大。要想改善人们的健康,将基因编辑真正地用在实处,我们不仅需要科学家的努力,还需要全社会一起努力。作者 | Sheldon 科学漫画工作室审稿 | MarvinP 加州大学洛杉矶分校研究员责编 | 高佩雯文章由腾讯科普“科普中国头条创作与推送项目”团队推出转载请注明来自科普中国注释:1. 在生物学中,A、T、G、C分别代表腺嘌呤(A)、胸腺嘧啶(T)、鸟嘌呤(G)、胞嘧啶(C)。生物学家常常把它们统称为碱基。在DNA分子中,碱基是成对出现的。其中,A和T组成一对,G和C组成一对。所以,一个DNA分子,就是由配对碱基组成的两行密码序列。并且,这两行密码序列保存的信息内容是完全等价的。当一个细胞分裂成两个细胞的时候,DNA分子就会以这两行密码序列为蓝本,进行自我复制,复制出两个一模一样的DNA分子,然后把它们分配给两个细胞。2. 一个DNA分子上包含大量的碱基序列,也就是包含大量的密码序列。其中一些密码序列读取以后,什么零件也造不出来,并不直接影响生物体的形成和发育,叫作非编码DNA。另一些密码序列会被细胞当作零件的图纸来读取,这些序列就叫作基因。细胞读取了基因图纸以后,就会参照图纸的说明,制造出相应的零件。其中每一种零件,就对应一种拥有特定功能的蛋白质。这些蛋白质有的变成了生物体的一部分,有的负责调控生物体的生长和发育。一个生物体的全部遗传信息,就叫作这种生物体的基因组。3. 3月1日,中科院神经所的杨辉团队在《科学》杂志上发表了一篇论文,用一种新技术(Genome-wide Off-target analysis by Two-cell embryo Injection)检测了几种基因编辑工具存在的缺陷。他们发现,CRISPR/Cas系统本身产生“错上加错”的情况并没有想象中的严重,但科学家改造它时给它额外添加的“剪辑编辑器”(BE3.0)会产生非常多的“错上加错”的结果。4. 本漫画已对基因编辑的过程做了大幅简化。实际过程跟科学家对“CRISPR/cas系统”的改造手段有关,也跟编辑基因时具体的需求有关。参考文献:1.王立铭,《上帝的手术刀》,浙江人民出版社。2.M. Eisenstein, CRISPR takes on Huntington’s disease, Nature 557, S42-S43 (2018).3.Dunbar CE et al., Gene therapy comes of age, Science, Vol. 359, Issue 6372, eaan4672.4.何东明, 中国学者在Science发表两篇论文,同时揭示碱基编辑器脱靶奥秘. http://www.zhishifenzi.com/news/biology/5322.html5.E. Zuo et al., Cytosine base editor generates substantial off-target single nucleotide variants in mouse embryos, Science 10.1126/science.aav9973 (2019).6.S. Jin et al., Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice, Science 10.1126/science.aaw7166 (2019).

















































