近年来,肠道细菌作为标志物诊断结直肠癌已达到令人满意的准确性。2021年5月,仁东医学研究院执行院长、同济大学生物信息系、同济大学附属第十人民医院朱瑞新教授团队等在Nature Communications期刊发表了题为:Identification of microbial markers across populations in early detection of colorectal cancer 的研究论文[1]。该研究实现了基于粪便微生物标志物的结直肠癌真正早筛,即结肠腺瘤(CRA)阶段的筛查(详情:Nature子刊:仁东医学助力结直肠癌早筛微生物标记物取得新进展)。同时,该工作也是紧抓我国政府颁布的《健康中国行动2030》关键:关口前移、癌前病变,靶向炎癌转化的标志性工作。
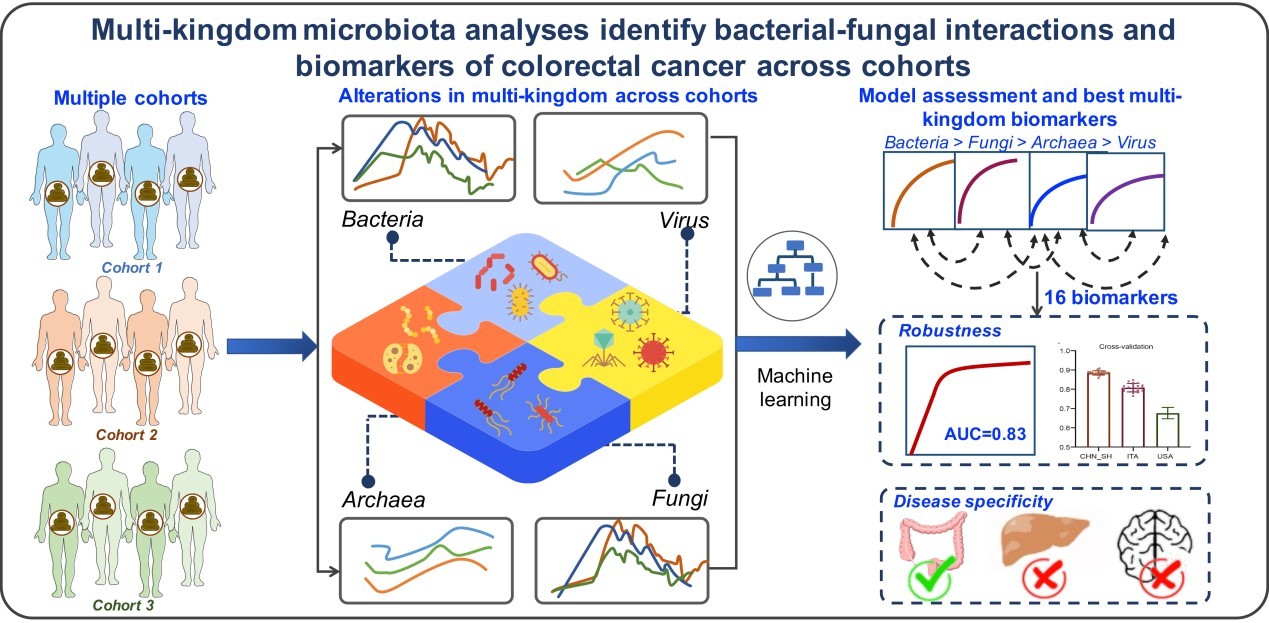
然而,除了细菌以外,真菌、古菌和病毒这些非细菌微生物群同样是肠道微生态的重要组成部分,这提醒了我们多界微生物在结直肠癌微生物群失调中的重要作用,然而,这一方向还没有得到有效探索。2022年1月27日,仁东医学研究院执行院长、同济大学生物信息系、同济大学附属第十人民医院朱瑞新教授团队和中山大学附属第六医院朱立新团队进一步联合上海交通大学医学院公共卫生学院王慧教授、中科院上海营养与健康研究所张国庆教授和复旦大学陈兴栋教授在 Nature Microbiology 期刊发表了题为:Multi-kingdom microbiota analyses identify bacterial–fungal interactions and biomarkers of colorectal cancer across cohorts (https://www.nature.com/articles/s41564-021-01030-7)的研究论文[2],开创性地实现了结直肠癌的多界微生物诊断。

该研究整合了全球8个国家/地区(中国、法国、德国、奥地利、意大利、美国、日本)的结直肠癌队列的共1368例标本的宏基因组数据,描绘了CRC相关的multi-kingdom微生物在全球的变化模式。系统评估了single-kingdom微生物针对结直肠癌的能力:发现基于single-kingdom微生物的诊断能力分别为细菌(27种细菌)>真菌(20种真菌)>古菌(20种古菌)>病毒(21种病毒)。随后该研究构建了multi-kingdom微生物的组合模型,发现multi-kingdom模型的诊断准确率优于single-kingdom模型,特别是包含细菌和真菌feature的模型表现更优,最终该研究确定了作为结直肠癌诊断标志物的高准确性的最小核心菌群(AUC=0.83):11种细菌、4种真菌和1种古菌。该multi-kingdom核心菌群的诊断效能在3个独立队列中表现出较强的鲁棒性(AUC分别为0.88,0.81和0.68),同时在非CRC疾病中验证了它们的疾病特异性,因此可作为基于粪便的无创结直肠癌诊断工具。此外,该研究还进一步探索了multi-kingdom微生物之间的相互作用以及微生物介导的功能变化,发现了细菌-真菌相互作用通过上调D-精氨酸、D-鸟氨酸以及刺激丁酸代谢通路促进结直肠癌发病的机制。同时,该研究还基于功能基因构建了CRC诊断模型,也具有较高的准确率(平均AUC为0.86)。
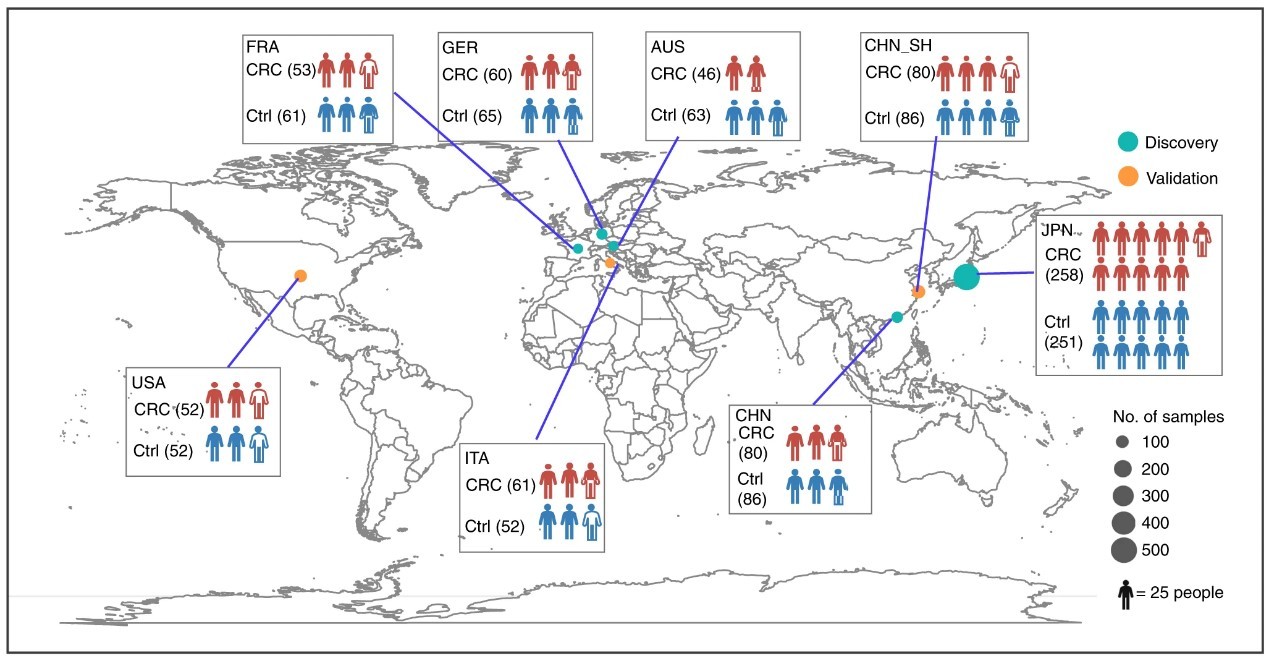
本研究纳入的CRC患者人群的全球分布
总的来说,该研究是迄今为止样本量最大、最全面的基于宏基因组测序的结直肠癌患者肠道微生物组的多中心队列研究,分析和绘制了全球八个结直肠癌人群队列的四界微生物组(古菌、细菌、真菌、病毒)图谱,确定了多界组合的微生物组比单界微生物组更能准确诊断结直肠癌,发现了结直肠癌特异的细菌-真菌互作新机制,并进一步提出了微生物及其相关功能基因作为治疗结直肠癌潜在靶点的可能性,为结直肠癌早期诊断和预后评估提供了基于微生物及其功能标志物的新方法和新思路。
仁东医学为本工作并列第一兼通讯单位。仁东医学研究院执行院长、同济大学生物信息系、同济大学附属第十人民医院朱瑞新教授、上海交通大学医学院公共卫生学院王慧教授、中科院上海营养与健康研究所张国庆教授、复旦大学陈兴栋教授和中山大学附属第六医院朱立新教授为该论文共同通讯作者。上海交通大学医学院公共卫生学院刘宁宁研究员、浙江大学医学院附属儿童医院焦娜研究员(原朱瑞新课题组博士生)、上海交通大学医学院公共卫生学院硕士研究生谭镜璁、上海市中医医院王子良研究员为该论文共同第一作者。整个项目的实施得到赵国屏院士的协调和指导。
仁东医学致力于液体活检无创诊断的开发应用,尤其近年在粪便微生物宏基因组检测层面已构筑完善的技术壁垒。人体粪便微生物的稳定检测非常依赖于宏基因组实验和分析流程的系统性和细节的准确性。通过与国内知名药企合作,仁东医学已经开发了宏基因组微生物检测的实验和分析一体化流程,并且应用于微生物与药物疗效的相关研究。
仁东医学的宏基因组微生物检测流程包括粪便样本采集、运输保存、核酸提取、文库构建、高通量测序、菌群群落结构分析、菌群功能分析和宿主-菌群互作分析和定制化报告解读等。产品验证工作表明,仁东医学在宏基因组微生物检测流程均达到较高的稳定性和准确性。稳定性方面,核酸提取的实验批次间,菌群群落结构一致性大于99%,文库构建的不同实验批次间,菌群群落结构一致性大于97%。另外,宏基因组文库在100-500ng不同起始量的实验条件下,菌群群落结构检测结果高度一致(大于99%),因此仁东医学在粪便宏基因组建库起始量最低可在100ng。准确性方面,通过与Zymo菌群标准品进行严格比较,仁东医学检测结果与标准品在物种水平的群落结构可以达到高度一致性87.14%,并且菌群丰度的检测极限可以达到0.001%,可以保证粪便样本真实菌群基本都被准确检测出来。
基于前期构建的粪便微生物宏基因组检测流程,针对本工作中确定的multi-kingdom核心菌群panel和相关的微生物功能基因,仁东医学正在开展相关菌群宏基因组检测产品设计,以实现更加简单、可及、价优和高性能的结直肠癌早筛早诊产品。
相关文献:
1.Wu Y, Jiao N, Zhu R, Zhang Y, Wu D, Wang A-J, Fang S, Tao L, Li Y, Cheng S et al: Identification of microbial markers across populations in early detection of colorectal cancer. Nature Communications 2021, 12(1).
2.Liu N-N, Jiao N, Tan J-C, Wang Z, Wu D, Wang A-J, Chen J, Tao L, Zhou C, Fang W et al: Multi-kingdom microbiota analyses identify bacterial–fungal interactions and biomarkers of colorectal cancer across cohorts. Nature Microbiology 2022.




 扫码下载APP
扫码下载APP

 科普中国APP
科普中国APP
 科普中国
科普中国
 科普中国
科普中国