背景
对 RNA进行测序一直以来都被认为是一种发现基因的有效方法,而且这种方法还被认为是对编码基因以及非编码基因进行注释的金标准。与以前的方法相比,大规模 平行RNA测序方法(massively parallel sequencing of RNA)极大增强了RNA测序技术的处理能力,使我们得以能够对转录组进行测序。在本文中即将介绍到的这两种RNA测序方法就能以前所未有的精度对转录组 进行分析。Trapnell小组使用的方法是一种名为Cufflinks的软件。这种软件能够随时发现小鼠生肌细胞(myoblast cell)内新出现的转录子,还能在细胞分化时对转录子表达水平进行监测,从而分析基因表达情况和剪接情况。Guttman小组也使用了与 Trapnell小组相类似的软件方法,不过他们使用的是另一种名为Scripture的软件。Scripture软件可以对源自三个小鼠细胞系的转录组 进行再注释(reannotate),从而对数百个最近新发现的lincRNA(large intergenic noncoding RNA)进行完整的基因模式注释。
虽然RNA测序技术已经出现了将近20年,但直到最近才开始构建克隆文库。对人类、小鼠以及其它重要模 式生物进行全长基因克隆构建的科研项目需要几年的时间才能够完成。但是有了最新的测序技术,我们将不再需要构建克隆文库,可以直接对cDNA片段进行测 序。我们现在可以只需要花费几天,仅用以往同类项目科研经费的很少一部分就能够得到一个比较满意的完整的细胞转录组。但是这种新技术也存在一点问题。不用 构建克隆,我们就无法知道哪一个“结果(mRNA或蛋白)”来自哪一个转录子。最近已经有人开始通过对已知的或者预测出来的转录子的短RNA序列进行测序 的方式来对基因表达和可变剪接进行分析研究。虽然这些研究可以得到很多信息,但是这种方法只能用于分析已知基因和对已知的可变连接区域进行分析。为了充分 利用RNA序列数据进行生物学研究,我们还应该能够重建转录子并且还要能够在不借助参考注释基因组信息的情况下对这些转录子的相对丰度进行精确的测量。
长链非编码RNA(lncRNA, lincRNA)数据库国内外长非编码RNA的研究刚刚兴起,种类大致包含以下几种。
(1)lncRNABase:提供miRNA调控长非编码RNA(lncRNA)、假基因(pseudogene)和环状RNA(circRNA)的互作信息和ceRNA调控网络。构建了最全面的包含了**14癌症类型(>6000个样本)Pan-Cancer(泛癌)**表达图谱和互作网络。这些调控互作网络信息是基于高通量的CLIP-Seq实验数据。
(2)LNCipedia: 对人类的长链非编码RNA的序列和结构全面的注释。
(3)**ChIPBase:**提供长链非编码RNA的表达图谱和转录调控的全面鉴定和注释。整合了高通量的RNA-seq鉴定的lncRNA及其表达图谱和ChIP-Seq实验技术鉴定的转录因子结合位点。
(4)oncoNcRNA:研究各类非编码RNA(lncRNA,miRNA, piRNA, tRNA和snoRNA等)在肿瘤中的表达模式和作用。这平台包含了64种癌症类型(>20000肿瘤样本和>900种癌症细胞系)的各类非编码RNA的表达和拷贝数变化数据(SCNA)。
(5)lncRNAdb: 提供有生物学功能的长链非编码RNA的全面注释。这是长链非编码RNA研究领域的大牛John mattick实验室构建的网站。
(6)LncRNADisease: 提供了文献报道的疾病相关的长链非编码RNA的注释。
(7)NONCODE:提供对长链非编码RNA的全面注释,包括表达和该团队开发的ncFANs计算机软件预测的lncRNA功能。这是非编码RNA研究的知名数据库,已经更新到第三版。
(8)NRED: 提供人和小鼠的长链非编码RNA在芯片数据的表达信息。这也是John mattick实验室构建的网站。
miR2Disease数据库miR2Disease数据库是一个人工注释的数据库,主要收录的是与人类疾病相关的microRNA信息,旨在为全世界的科研工作者提供一个全面的与疾病相关的RNA数据资源平台。
miR2Disease数据库是免费使用的,可以登陆网页www.miR2Disease.org了解更多信息。
miRBase数据库是一个提供包括miRNA序列数据、注释、预测基因靶标等信息的全方位数据库。作为存储miRNA信息最主要的公共数据库之一,miRBase提供了便捷的网上查询服务,允许用户使用关键词或序列在线搜索已知的miRNA和靶标信息。但它还存在着一些不足的方面,比如植物预测靶标的描述比较模糊、茎环结构显示方式不够直观、特定miRNA相关功能描述的缺乏和对具反馈调节功能miRNA标注的缺乏等问题。希望在不久的将来,随着miRNA研究的不断深人,miRBase也建设成一个功能强大的全方位综合性数据库。
数据搜集以及数据库内容miR2Disease数据库中的记录主要提供的是microRNA失调与人类疾病的关系。他们使用诸如“microRNA disease”、“miRNA disease”、“microRNA cancer”、“miRNA cancer”等关键词搜索了PubMed数据库。最新版的miR2Disease数据库是在工作人员浏览了600多篇文献的基础上总结出来的,共收录有299个人类microRNA信息、94种人类疾病信息以及1939条microRNA与疾病之间的关联信息。
在miR2Disease数据库中他们使用的是由专业的医学词汇(即UMLS系统,参见http://diseaseontology.sourceforge.net/)来命名的疾病名称,这样便于用户搜索和分析资料。数据库中的每一条记录都包含详细的microRNA与疾病之间的关联信息、microRNA ID号、相关疾病名称、患病时microRNA表达模式(上调还是下调)介绍、microRNA表达模式相关试验技术介绍(芯片、northern blot、qRT-PCR等)、microRNA靶基因介绍(这些基因都是参阅相关文献或者直接从TarBase数据库中查阅而来)。
miR2Disease数据库使用的界面非常人性化,用户可以很方便地使用microRNA ID号、相关疾病名称或者靶基因名称进行查询操作,而且还提供了许多外部microRNA数据库的链接,例如microRNA序列及注释信息数据库miRBase、经过试验验证的microRNA靶基因数据库TarBase,以及microRNA功能计算机预测数据库TargetScan、miRanda和PicTar。
此外,miR2Disease数据库还提供与NCBI PubMed中参考文献之间的超链接及该文献在PubMed中的ID号和完整的引文介绍。在最新版本的miR2Disease数据库中,有1/7的记录是有关microRNA失调导致人类疾病相关信息的。
用户界面介绍搜索页面miR2Disease数据库可以通过搜索引擎在库中查询microRNA——疾病相关信息,用户可以通过microRNA ID号、相关疾病名称或者靶基因名称进行查询。
miR2Disease数据库还提供一种模糊查找功能,用户即使不知道疾病的确切名称,也可以借助该功能通过医学疾病参考词汇进行查询。
用户输入一个查询关键词之后,系统就会在数据库中搜索出所有包含该关键词的记录,这些记录会以“疾病树(disease tree)”的形式展现出来,其中既包含了疾病本身,也包含其“祖先节点(ancestor node)”和所有的亚类。在“疾病树(disease tree)”中包含查询关键词的部分会以高亮粗体的方式显示出来,每一个包含有microRNA——疾病相关信息的部分也会给出超链接,用户通过这些超链接就可以很轻松地获得与该疾病相关的所有microRNA的信息。在搜索结果页面,点击每一条记录末尾处的‘more . . .’按钮可以了解到更多相关的信息(如图)。
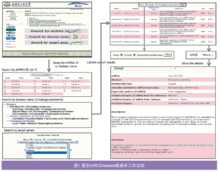
与使用疾病名称进行搜索差不多,使用microRNA名称进行搜索也有可能出现混淆与混乱,因为大部分的原始文献都不会给出某一个microRNA家族中每一个具体microRNA的详细信息及名称。例如在很多文献中提到的“let-7”是与腺瘤相关的,但还有一些文献中则报道说在乳腺癌患者体内let-7a-3的表达水平会降低。因此,通过模糊查询功能,用户哪怕在不清楚microRNA准确名称的情况下也能在miR2Diesease数据库中查询到相关信息。
有时使用一个microRNA名称可以查到好几个ID号,用户可以根据自己的兴趣做进一步选择。图1也给出了使用miRNA ID号进行查询的简要流程图。
在miR2Disease数据库中,靶基因可以被分为三类,即原始参考文献中报道的靶基因;TarBase数据库(该数据库收录的都是经过试验验证的数据)报道的靶基因;以及使用计算机软件(例如Miranda、TargetScan、PicTar)预测出的靶基因。用户可以使用第一和第二种靶基因进行搜索,目前还不支持使用第三种靶基因进行搜索,不过今后的版本肯定能支持该功能。
microRNA与疾病的关系在搜索结果页面和详细介绍页面都有显示(图1)。而且,miR2Diesease数据库还提供过滤功能,用户可以选择只显示microRNA与疾病关系的相关信息。
提交页面miR2Disease数据库还设有用户提交页面,供用户上传目前miR2Disease数据库中还没有收录的有关microRNA与人类疾病关系的相关数据资料。一旦miR2Disease数据库评审委员会审核通过用户提交的信息,数据库就会将该信息收录进来,在接下来的新版本中公布。miR2Disease数据库会每月更新一次。
讨论越来越多的证据表明,某种microRNA时空特异性的表达对于细胞正常的发育分化过程起到了至关重要的作用,而microRNA的异常表达则与许多人类疾病有关。为了给研究microRNA与人类疾病关系的科学家们提供一个集中的相关数据资源,研究人员有了建立miR2Disease数据库的想法。他们主要关注microRNA失调与人类疾病的关系。
miR2Disease数据库不仅仅是一个供用户查询信息的数据库,同时它也是一个可以上传资料,供大家交流信息、分享成果的平台。

失调的microRNA在miR2Disease数据库收录的条目中大约有1/7的microRNA失调导致疾病发生,它们会引起癌症、代谢性疾病、心血管疾病等等。例如Ma等人报道的miR-10b高表达抑制了HOXD10基因的翻译,引起RHOC这种促转移基因(pro-metastatic gene)高表达,从而导致乳腺癌扩散、转移。再如Huang等人发现miR-373和 miR-520c的明显上调抑制了CD44表达,因而刺激了乳腺癌细胞的转移。Meng等人则发现癌基因——促分裂原活化蛋白激酶激酶(mitogen-activated protein kinase kinase kinase 8, MAP3K8)是miR-370的靶基因,miR-370下调会导致MAP3K8基因表达水平升高,引起胆管癌(cholangiocarcinoma)。还有报道miR-375通过调控肌侵蛋白(myotrophin)的表达能控制胰岛素分泌,miR-375上调能抑制胰岛素释放。还有研究表明miR-1和miR-133下调与HCN2/HCN4再表达(reexpression)和肥大心肌细胞电重构(remodeling)等有关。
miR2Disease数据库中的信息还可以用来研究不同疾病之间的关系,他们为此专门设立了一个双向网络(bipartite network),以描述85种microRNA和32种癌症相关疾病之间的因果关系(图3)。

可能导致microRNA失调的机制首先,microRNA都是位于与疾病有关的基因座内,例如杂合子缺失的微小区域(minimal regions of loss of heterozygosity)、微小扩增区域(minimal amplicon)或断裂位点(breakpoint cluster region)等脆性位点区域。比如miR-15和miR-16都位于人染色体13q14区域,该区域在超过半数的B细胞性慢性淋巴细胞性白血病(B-cell chronic lymphocytic leukemia,B-CLL)患者中都出现缺失。因此,大多数的B-CLL患者(68%)中这两个基因也都有缺失或表达下调。相反,miR-17-92多顺反子位于B细胞淋巴瘤基因扩增区域,因此会过表达。
其次,microRNA失调是由异常的表观遗传学改变(epigenetic patterns)所致。例如DNA异常甲基化、组蛋白异常修饰等等。比如,在正常情况下,人类正常组织中let-7a-3基因的启动子区域是高度甲基化的,但是在肺癌组织中该区域则是低甲基化的。这种启动子低甲基化状态会引起let-7a-3这种癌基因激活,引发肺癌。此外,异常的高甲基化会导致乳腺癌患者体内miR-9-1失活。
最后,microRNA失调可能是由参与microRNA生成的酶功能异常所致。比如,Otsuka等人发现的miR-24和miR-93可以针对病毒L蛋白(large protein)和P蛋白(phospho protein)基因。在Dicer1缺陷的细胞中,缺乏miR-24和miR-93会增强VSV病毒的复制效率。在miR2Disease数据库中,上述这些信息都位于“分析类”中,可以从网站主页上链接进入。
除了上述内容之外,miR2Disease数据库中还收录有关哪些导致疾病的基因变异位点能通过干扰microRNA的靶向效应来影响细胞功能的条目。例如,let-7:Hmga2、miR-148a:HLA-G和miR-433:FGF20之间的相互靶向作用消失分别与肌瘤(myoma)、哮喘(asthma)以及帕金森氏症(Parkinson’s disease)相关。
Chen等人还发现CCND1 mRNA通常都会受到miR-16-1的调控,而在外套细胞淋巴瘤(mantle cell lymphoma)细胞中,CCND1基因的截短突变体则不受miR-16-1的调控,因为该突变体基因的mRNA上没有miR-16-1的结合位点。总之,microRNA不能对靶基因进行调控(microRNA与mRNA之间失去了相互作用的能力或不能互相结合等)是microRNA致病的一条新机制。
综上所述,miR2Disease数据库为人们提供了一个有关microRNA失调与人类疾病关系方面的综合网上数据资源平台。有理由相信miR2Disease数据库会成为生命科学和生物学界研究工作者的好帮手,能帮助他们进一步了解microRNA失调与人类疾病之间的关系。
未来的发展方向由于miR2Disease数据库的主要工作是“数据收集与整理、储存”,因此,研究人员目前主要是使用一些诸如“microRNA disease”、 “miRNA disease”、 “microRNA cancer”、“miRNA cancer”等关键词在PubMed数据库中进行有关microRNA与人类疾病关系方面的资料搜集整理工作。虽然到目前为止研究人员已经从600多篇文献中搜集到了约2000条microRNA与人类疾病的关系并收录入miR2Disease数据库中,但是由于存在上述建库方法学上的局限性,因此,miR2Disease数据库目前还缺乏全面性与系统性。所以他们计划采用两种新的策略来改进采集数据的方法。第一条策略是将使用文本挖掘工具(text-mining tool)来帮助对PubMed数据库的摘要进行预检索(prescreen),以此来扩大初步搜索的范围。第二条策略是将重点关注美国国家医学图书馆(National Library of Medicine)建立的MeSH词汇表更新情况。1
毫无疑问,上述这两条策略将丰富miR2Disease的数据量,扩大数据库的覆盖范围,相信在即将发布的新版miR2Disease数据库中,用户就会看到这些改变了。
值得注意的是不是所有的microRNA与疾病之间都有非常明确的关系,miR2Disease数据库中有一些记录比其它的记录有更明确的microRNA——疾病关系。




 扫码下载APP
扫码下载APP

 科普中国APP
科普中国APP
 科普中国
科普中国
 科普中国
科普中国