
图片来源:https://www.jiemian.com/article/506742.html
人生路上常常遇到分叉路口,在这个选择的一瞬间我们的命运将会改变,选择A,可能会成为制作舌尖上的美食的大厨;选择B,可能成为此刻在电脑前码代码的我。
众所周知,多能干细胞具有分化成不同类型的子细胞的多能性。而对于这些干细胞来说,在它分化的路上也会遇到“向左走”还是“向右走”的难题。此前大量的研究发现,**这个选择的关卡是由一些关键的转录因子调控的,**但是这些转录因子如何决定干细胞的分化进程还并不完全清楚。 近年来,随着计算生物学的发展特别是单细胞组学的迅猛发展,科学家有机会通过综合数学和统计模型并结合组学数据来研究细胞命运的发展。
造血干细胞是研究细胞命运选择的经典模型,在脊椎动物的骨髓里,有限的造血干细胞可以分化出体内各类成熟的血细胞,这个作用受到转录因子和DNA元件严密的平衡调控。
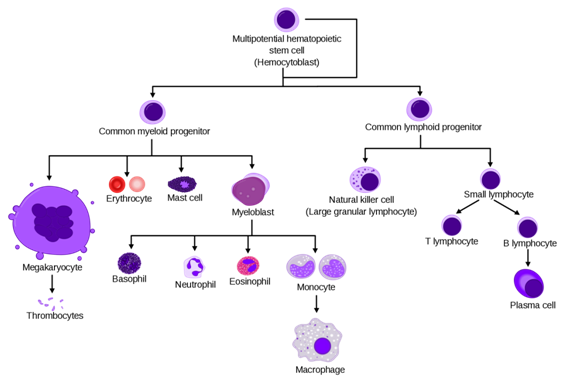
图 | 造血干细胞分化的经典模型
(多能造血干细胞分化成髓细胞和淋巴细胞前体细胞,再进一步分化成粒细胞、红细胞、淋巴细胞等。)(图片来源:维基百科“造血干细胞”词条)
尽管此前的研究通过测量RNA的表达量来量化不同子细胞中的转录因子的变化,但大家都知道,RNA的表达量并不等于蛋白质的表达水平。目前,来自牛津大学Weatherall研究所计算生物学中心的Morrissey博士课题组和加拿大渥太华干细胞研究中心的Brand教授课题组合作在Cell Stem Cell发表了通过单细胞蛋白质组学的方法,测定造血干细胞转录因子共表达谱系的时间特异性变化,首次直接提供了造血干细胞命运决定的蛋白质表达数据。

图片来源:Cell Stem Cell 网站截图
研究人员首先对多能干细胞(CD34+ HSPC)分化为红细胞的完整进程(共22天,包括各个进程的红细胞)定时取样(每隔两天), 在每个时间点对每个单细胞进行标记,通过27种抗体联合标记对应的蛋白质,再通过质谱流式细胞术(CyTOF)同时测定多种蛋白质的表达,这样科学家们就可以比较从多能造血细胞到红细胞的分化过程中13个连续时间点的关键蛋白质表达水平。
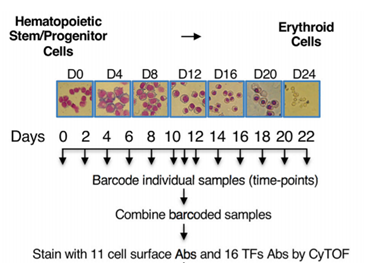
图 | 实验过程示意图(图片来源:论文)
研究人员首先使用最近邻居算法 (KNN Algorithm)将数据可视化,通过构建蛋白质表达水平相似的细胞之间的连接网来找到不同的细胞分化路径,发现了干细胞分化为红细胞和巨型吞噬细胞的两个方向的重要蛋白。为了确定具体的细胞亚型,研究人员根据CyTOF数据的统计学特性,开发了新的分类算法 (Clustering Method)—负二项分布统计法,这个算法可以根据单细胞蛋白质表达的水平进行分类,确定细胞亚型,从而比较不同细胞亚型的蛋白质水平。
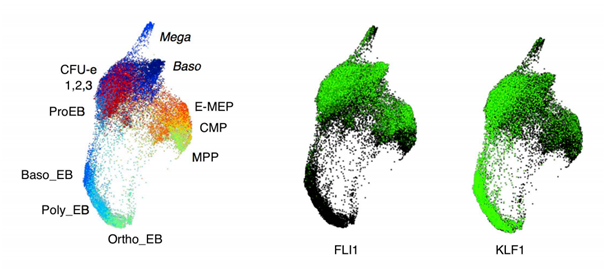
图|FLI1和KLF1在不同细胞中的表达(图片来源:论文)
通过开发的新算法,研究人员发现,从干细胞到红细胞/巨型吞噬细胞的分化路径中,转录因子KLF1和FLI1皆有表达,但两者的表达水平不同,当KLF1的表达水平高于FLI1时,分化导向为红细胞,反之,为巨型吞噬细胞,研究人员还通过实验证实了这一分化路径。
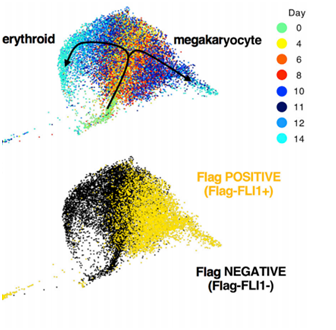
图 | 红细胞和巨核细胞分化路径图
(在某一个时间点后细胞走向了两条完全相反的分化路径,FLI1过表达的细胞组中,绝大部分的细胞都分化为巨型吞噬细胞。)(图片来源:论文)
这是科学家们首次通过测量蛋白质表达水平完整地展示了造血干细胞分化过程中连续时间的转录因子表达水平,本研究中开发的新算法为干细胞命运决定的分析提供了新思路,该研究为更好的理解干细胞的分化的命运决定提供了数据基础。
与此同时,该研究也为相关的血液疾病(如急性白血病)的治疗提供了蛋白组学的信息,这些时空水平的蛋白质信息也对后续的药物研发提供了理论基础。
本文作者: 程谦博士(论文共同第一作者),先后毕业于英国牛津大学、剑桥大学,现为英国牛津大学Weatherall分子医学研究所计算生物学中心博士后研究员,合作导师为Edward Morrissey博士,通过开发和应用数学与统计模型研究组织内的干细胞命运。
课题组主页:https://www.imm.ox.ac.uk/research/units-and-centres/mrc-wimm-centre-for-computational-biology/groups/quantitative-biology-of-cell-fate-and-tissue-dynamics.
参考文献:
-
Palii, C.*, Cheng, Q.*, Gillespie, M., Shannon, P., Mazurczyk, M., Napolitani, G., Price, N.D., Ranish, J.A., Morrissey, E., Higgs, D.R., Brand, M., Single-cell proteomics reveal that quantitative changes in co-expressed lineage-specific transcription factors determine cell fate. Cell Stem Cell, online
-
Palii, C.G., Pasha, R., and Brand, M. (2011). Lentiviral-mediated knockdown during ex vivo erythropoiesis of human hematopoietic stem cells. J. Vis. Exp. 2813.
-
Kester, L., and van Oudenaarden, A. (2018). Single-cell transcriptomics meets lineage tracing. Cell Stem Cell 23, 166–179.
-
Spitzer, M.H., and Nolan, G.P. (2016). Mass cytometry: single cells, many features. Cell 165, 780–791.




 扫码下载APP
扫码下载APP

 科普中国APP
科普中国APP
 科普中国
科普中国
 科普中国
科普中国