医学图像分析(Medical Image Analysis)是医学术语,指综合医学影像、数学建模、数字图像处理与分析、人工智能和数值算法等学科的交叉领域。
医学图像分析简介20世纪70年代,X线图像在临床广泛应用,医生借助学习得到的知识和实践积累的经验,解读病人的X线照片所反映的解剖结构和病生理信息。但是,这种人工解读方式,往往依赖于医生个人的经验、知识和情绪,且效率较低。随着计算机的发展和数字化仪器的出现,人们开始把X线胶片的模拟图像转变为数字图像存储和传输;另外,计算机技术的逐步成熟也提供了相当的计算能力。研究者开始尝试把医学模拟图像转化为数字图像,开展了计算机辅助诊断(computer aided diagnosis,CAD)的初步研究,试图在一定程度上辅助医生判读医学图像,排除人为主观因素,提高诊断准确性和效率。
医学临床需求也推动着医学图像分析的发展。多排螺旋CT能够在极短的时间内采集到真正的各向同性体素数据并重组成三维数据,MR心脏图像是时间加三维的四维数据,一次完整的心脏检查结果包括在心动周期上、各个时刻、多个层面上的数百张二维图像。数据采集的高维化,导致依靠传统的二维表达已经很难理解如此海量的信息,更谈不上有效率地判读和分析。医学图像分析的研究的对象也日益广泛,不再局限于过去具有明显诊断特征的病种,开始扩展到多种不同器官、解剖形态、功能过程的图像,试图利用自动精确定量的计算机辅助图像分析,帮助临床医生和研究者高效准确地处理海量图像信息。在成像硬件的推动下,从20世纪90年代中期到现在,医学图像分析在理论方法和应用上都取得了长足的进步。
理论分割医学图像的感兴趣区分割研究,是医学图像分析中最重要的基础,准确、鲁棒和快速的图像分割,是定量分析、三维可视化等后续环节之前的最重要步骤,也为图像引导手术、放疗计划和治疗评估等重要临床应用奠定了最根本的基础。早期医学图像分割研究,主要是直接应用图像处理的经典方法,如边界提取和基于灰度的区域增长算法等。后来开始针对不同的解剖位置和成像模态,设计基于成像设备、成像参数和解剖结构等先验知识的专用图像分析算法,如利用多参数的MR图像、决策树和统计聚类技术来分割脑组织的白质和灰质。随着图像分析和计算机视觉理论方法的不断丰富,医学图像感兴趣区分割也出现了很多新突破。参数动态轮廓线模型,(active contour model)开创性地提出了高层模型指导、底层图像特征驱动的图像分割思想,通过最小化模型的内外部能量得到分割结果,在随后20多年中得到研究者的广泛关注和不断发展 。动态表现模型(active appearance model) ,动态形状模型(active shape model) 都是这一类的代表方法。Osher和Sethian提出的水平集(1evel set)方法 ,利用数值方法模拟各种曲面演化,后来作为一种数值模拟和分析的方法,被广泛运用于曲线或曲面的运动跟踪,在医学图像分割中也得到广泛应用。但目前大多数分割算法对医学图像成像参数和曲线、曲面的初始位置较敏感,没有人工参与,难以做到鲁棒分割不同设备乃至同种设备、不同参数的医学图像。
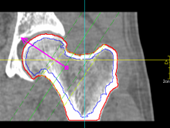
图1. 医学图像分割
图像配准和信息融合早期医学图像的配准目的,是为了把显示人体不同信息(结构信息和功能信息)的医学图像放到统一坐标系中显示。随着脑部结构形态学分析研究的进步,出现了同病人不同时期及同种疾病的病人群体图像数据配准,以及标准图谱到特定图像数据集配准研究。为了消除成像过程中对象运动的影响,图像配准也是时序图像分析的第一步。寻找物体在不同影像中对应像素点的关系是医学图像配准的基本任务。通过寻找适当的空间变换,使图像数据达到空间位置上的定位和配准,进而进行图像融合。图像配准方法可以分为基于外部特征和基于内部特征两类。使用外部特征的通过人工设置的标记点实现配准,如立体框架定位、皮肤标记法等;而基于内部特征方法通过寻找图像内部解剖结构上的特征点或外部轮廓和表面的对应关系实现配准。互信息量是随机变量统计相关性的测度,在医学图像配准中得到广泛应用 。由于最大互信息方法不需要假设不同成像模式下的图像灰度的相关性,也无需对图像进行分割和预处理,非常适合三维多模医学图像配准,具有精度高、鲁棒性强的特点。目前,医学图像配准技术和分割技术一样,出现了很多成熟的方法和软件,是医学图像分析中的基本技术之一。
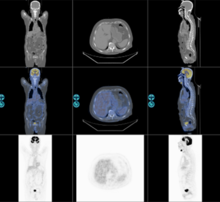
图2. 医学图像配准融合
可视化可视化(visualization)在医学图像分析中扮演重要角色。医学图像可视化技术是指利用从实验中获得的、扫描器测得的、计算模型合成的医学图像数据,重建三维图像模型,并进行定性定量分析,为用户提供具有真实感的三维医学图像,使人们更清楚地认识蕴涵在体数据中的复杂结构,便于医生多角度多层次地观察和分析,并且能够使医生有效参与数据处理与分析的过程。医学图像可视化技术,按照绘制过程中数据描述方法的不同,通常分成两大类一面绘制和体绘制。面绘制技术是指对体表面重建,即从切片数据提供的三维数据场中抽取等值面,然后用传统图形学技术实现表面绘制。面绘制可以有效地绘制出物体表面,但缺乏对物体内部信息的表达。体绘制则是以体素作为基本单元,直接由切片数据生成三维物体图像,表示物体内部信息,但是计算量很大。此外,将那些融合了这两种技术特点的重建算法归纳为第3大类——混合绘制技术。
体绘制技术不需要构造中间对象,直接由三维数据本身来重建物体。三维数据中的各个数据作为一个表示实体的基本单元——体素,每个体素都有颜色、不透明度、梯度等相应属性。体绘制的基本原理是模拟光线穿越半透明物质时能量的累积变化,即光线传输理论。具体说来,首先对每个体素赋以不透明度和颜色值(R、G、B);再根据各个体素所在点的梯度以及光照模型计算相应体素的光照强度;然后根据光照模型,将投射到图像平面中同一个象素点的各个体素的不透明度和颜色值组合在一起,生成最终结果图像。体绘制技术的典型算法有光线投影法、足迹法、错切变形法、基于硬件的三维纹理映射、频域体绘制法、基于小波的体绘制法等。
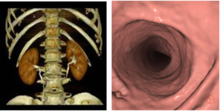
图3. 医学图像可视化
时序图像的功能分析医学图像分析的目的是得到生理过程的定量信息,为临床诊疗提供更充分的依据。过去的医学成像主要集中在解剖结构成像和形态分析上。近年来,医学设备成像速度的飞跃使对生理过程的动态观察成为可能。例如:在脑功能成像领域,Functional MRI开始广泛应用;核医学PET(positron emission tomography)和SPECT(single photon emission computer tomography),在脑功能和心功能动态成像方面也得到广泛的应用;MR心功能成像方面,标记MR技术(Tagging)和相位对比MR成像技术,在物理上为图像定量分析心功能提供了依据;另外,三维超声的出现,提供了实时观察心室和瓣膜运动的手段。以上设备都能生成产生时序的二维或三维医学图像,反映了生理过程的动态功能信息。相应的医学图像分析也由图像处理范畴的图像分割、配准技术,向以医学图像为媒介,精确、定量地分析器官的功能和生理过程发展。
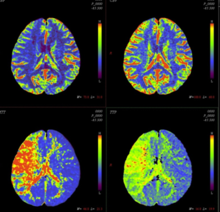
图4. 脑部灌注分析
功能拓展及应用基于内容的图像检索(content based image retrieval)在自然图像和视频检索方面取得一定进步,通过提取输入图像的特征,在图像数据库快速搜索特征空间中邻近的类似图像。随着医学数据的海量化趋势加剧,近几年来基于内容的医学图像检索的研究与应用正形成热点。图像检索系统可以帮助医生在海量数据库中快速寻找具有类似病理特征并已确诊的医学图像,提高疾病诊断的准确率。医学图像检索有以下难点:首先,不同成像设备得到的医学图像内容特点差异巨大,很难用统一算法自动分析和提取特征;其次医学图像的分类涉及专业知识,譬如区分不同种类的脑部肿瘤图像,必须通过某种形式来结合专科影像学专业知识;再次,现代的医学图像多为三维或者更高维,这也给特征提取和检索算法提出了新的挑战。上述困难使目前图像检索研究主要集中在针对特定成像设备和特定解剖位置,或疾病种类的医学图像。目前,大部分研究止于理论研究,只有及少数图像检索系统报告了临床评估的表现和实际应用。
扩展阅读陈武凡, 秦安, 江少峰, 冯前进, 郝立巍. 医学图像分析的现状与展望, 《中国生物医学工程学报》,2008年第27卷第2期 175-181页。
汤敏, 陈峰, 陶玲. 医学图像可视化及加速技术的研究进展和趋势, 《中国组织工程研究与临床康复》,2011年第15卷第4期 741-744页。
J Gee, M Reivich, R Bajcsy (1993). "Elastically Deforming a Three-Dimensional Atlas to Match Anatomical Brain Images". Journal of Computer Assisted Tomography 17 (1): 225–236.
MR Sabuncu, BT Yeo, K Van Leemput, B Fischl, P Golland (June 2010). "A Generative Model for Image Segmentation Based on Label Fusion". IEEE Transactions on Medical Imaging 29 (10): 1714–1729.
Cootes TF, Taylor CJ, Cooper DH, and Graham J (1995). "Active shape models-their training and application". Computer vision and image understanding (San Diego: Academic Press, c1995-) 61 (1): 38–59.
Cootes, T.F. and Edwards, G.J. and Taylor, C.J. (2001). "Active appearance models". Pattern Analysis and Machine Intelligence, IEEE Transactions on (IEEE) 23 (6): 681–685.
R. Goldenberg, R. Kimmel, E. Rivlin, and M. Rudzsky (2001). "Fast geodesic active contours". IEEE Transactions on Image Processing 10 (10): 1467–1475.
Karasev, P., Kolesov I., Chudy, K. Vela, P., and Tannenbaum, A. (2011). "Interactive MRI segmentation with controlled active vision". Proceedings of the Conference on Decision and Control.
Lisa Gottesfeld Brown (1992). "A survey of image registration techniques". ACM Computing Surveys (CSUR) 24 (4): 325–376.
J. Maintz and M. Viergever (1998). "A survey of medical image registration". Medical Image Analysis 2 (1): 1–36.
J. Hajnal, D. Hawkes, and D. Hill (2001). Medical Image Registration. Baton Rouge, Florida: CRC Press.
Barbara Zitová, Jan Flusser (2003). "Image registration methods: a survey". Image Vision Comput. 21 (11): 977–1000.
J. P. W. Pluim, J. B. A. Maintz, and M. A. Viergever (2003). "Mutual information based registration of medical images: A survey". IEEE Trans. Med. Imaging 22 (8): 986–1004.
C. J. Twining and T. Cootes and S. Marsland and V. Petrovic and R. Schestowitz and C. Taylor. "A unified information-theoretic approach to groupwise non-rigid registration and model building". Proceedings of Information Processing in Medical Imaging 2005.
M. Sabuncu and S. K. Balci and M. E. Shenton and P. Golland (2009). "Image-driven Population Analysis through Mixture Modeling". IEEE Transactions in Medical Imaging 28 (9): 1473–1487.
K.J. Friston, W.D. Penny, C. Phillips, S.J. Kiebel, G. Hinton, and J. Ashburner (2002). "Classical and Bayesian Inference in Neuroimaging: Theory". NeuroImage 16 (2): 465–483.
Yong Fan, Nematollah Batmanghelich, Chris M. Clark, Christos Davatzikos, the Alzheimer’s Disease Neuroimaging Initiative, Spatial patterns of brain atrophy in MCI patients, identified via high-dimensional pattern classification, predict subsequent cognitive decline. (2008). "Spatial patterns of brain atrophy in MCI patients, identified via high-dimensional pattern classification, predict subsequent cognitive decline". NeuroImage 39 (4): 1731–1743.
Y. Wang and Y. Fan and P. Bhatt P and C. Davatzikos (2010). "High-dimensional pattern regression using machine learning: from medical images to continuous clinical variables". Neuroimage 50 (4): 1519–35.
Benoît Magnin and Lilia Mesrob and Serge Kinkingnéhun and Mélanie Pélégrini-Issac and Olivier Colliot and Marie Sarazin and Bruno Dubois and Stéphane Lehéricy and Habib Benali (2009). "Support vector machine-based classification of Alzheimer's disease from whole-brain anatomical MRI". Neuroradiology 51 (2): 73–83.
N.K. Batmanghelich and B. Taskar and C. Davatzikos (2012). "Generative-discriminative basis learning for medical imaging". IEEE Trans Med Imaging 31 (1): 51–69.
Glenn Fung and Jonathan Stoeckel (2007). "SVM feature selection for classification of SPECT images of Alzheimer's disease using spatial information". Knowledge and Information Systems 11 (2): 243–258.
本词条内容贡献者为:
李宏 - 讲师 - 东北大学中荷生物医学与信息工程学院张耀楠 - 教授 - 东北大学中荷生物医学与信息工程学院




 扫码下载APP
扫码下载APP

 科普中国APP
科普中国APP
 科普中国
科普中国
 科普中国
科普中国