人类的可遗传疾病有千千万万种,不知道你们有没有想过,科学家到底是通过何种神通的本领寻找到这些遗传疾病背后的致病基因的呢?今天就让我们一起来聊聊科学家寻找疾病遗传变异的进阶之路。
虽然人类可遗传疾病的种类非常多,但是根据疾病致病基因的数量,可以将这些可遗传疾病分为两大类:单基因遗传病和多基因遗传病。
单基因疾病
顾名思义,单基因遗传病是由一对等位基因的变异引起的,因此符合典型的孟德尔遗传规律。按照遗传方式的不同,单基因疾病又可以分为常染色体显性遗传病(比如亨廷顿舞蹈症)、常染色体隐性遗传病(如白化病)、X连锁显性遗传病(如佝偻病)、X连锁隐性遗传病(如红绿色盲)、Y连锁遗传病(如耳廓长毛症)五种。据不完全估计,目前已经发现超过6600种单基因遗传病,并且这一数字每年还在不断增加中。
单基因遗传病的定位研究相对来说要简单一点,系谱分析是目前最为常用的研究方法。一个足够大的家系有时就足以判定单基因疾病的遗传方式,再借助如今的高通量的测序技术测定出患者可能的致病基因序列,通过与数据库中的序列进行比对,从而能够锁定单基因疾病的基因缺陷。目前临床上对1000多种单基因疾病的发病机制研究的已经比较清楚,临床上也有了相对成熟的检测手段。
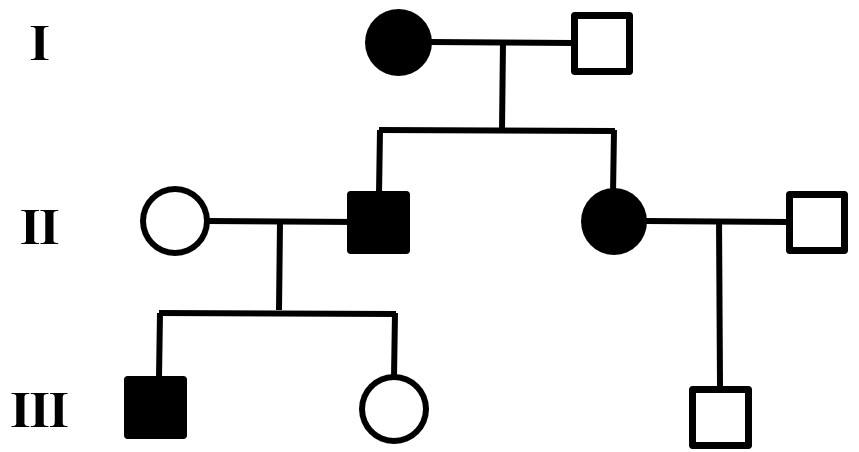
多基因疾病
相对于“一个基因,一种疾病”的单基因疾病,多基因遗传病的研究之路则更为坎坷。原因在于,多基因遗传病通常由成百上千的基因变异与环境因素(如饮食习惯,睡眠,压力和吸烟等)共同作用产生,虽然有家族聚集倾向,但在一个家系中却并没有明显的孟德尔遗传方式。事实上,虽然我们人类有数千种单基因疾病,但它们仍然是比较少见的。相反,我们生活中常见的很多疾病,比如高血压、糖尿病、哮喘、抑郁症、精神分裂症和先天性心脏病等都是多基因遗传病。
接下来我们就以抑郁症为例,看看科学家是如何寻找复杂疾病的遗传变异的。
什么是抑郁症?
抑郁症是以显著而持久的心境低落为主要临床特征的精神障碍。它的主要症状包括情绪低落,兴趣减低、悲观、思维迟缓、缺乏主动性、饮食和睡眠差等,严重者会反复出现自杀想法和行为。我们所熟悉的张国荣和乔任梁等多位明星正是由于不堪忍受抑郁症的折磨而自杀,世界上著名的喜剧大师卓别林和“憨豆先生”也曾经饱受抑郁症的折磨。调查显示,抑郁症的终生患病率(在一生当中得过抑郁症的患者占总人口的比率)约为6.8%,照此计算,超过9500万中国人一生当中可能会经历这样一场“精神感冒”。

抑郁症是受遗传与环境因素共同作用的结果。
研究表明抑郁症的发生与一些社会和环境因素密切相关。这些因素包括儿童期的性、身体或情绪虐待,消极生活事件的经历,如疾病、失去亲人或朋友和失业等,以及不良生活方式(例如饮酒、吸烟、高脂肪或高糖饮食以及缺乏运动等)。除了环境因素,遗传也是重要的抑郁症风险因素。比如,很早以前,人们就已经认识到抑郁症具有在家族内聚集的遗传倾向,并且抑郁症患者的一级亲属(父母、子女以及兄弟姐妹)的患病风险比普通人增加了2-3倍,而针对抑郁症的双胞胎研究和领养研究表明遗传因素对抑郁症的影响大约能占到30–40%。
那么科学家是怎么去寻找抑郁症这种复杂疾病的致病基因的呢?
候选基因研究
在全基因组测序普及之前,科学家通常采用候选基因法来研究抑郁症的致病基因。在这种模式下,研究者基于“经验”(例如已发表的研究论文以及动物模型实验),提出人类抑郁行为可能与大脑神经活动相关,进而锁定了一系列可能会影响到神经活动的基因,以此作为候选基因,通过大量实验及数据分析验证这些候选基因是否与抑郁症发生相关。通过这种方法,在过去20多年,研究者找到了很多与抑郁症相关的明星基因。例如编码血清素转运体的基因SLC6A4、编码脑源性神经营养因子的基因BDNF以及编码大脑特异性的色氨酸羟化酶的基因TPH2等。然而,由于大多数候选基因研究的样本量比较小,加上学术杂志又比较偏向发表有阳性结果的论文(出版偏倚),导致这些候选基因研究所报道的结果难以重复。而在2019年,《美国精神病学杂志》发表了一篇论文重新评估了18个曾被广泛研究的抑郁症候选基因(包括SLC6A4、BDNF和TPH2等)与抑郁症的关系,发现这18个抑郁症候选基因可能跟抑郁症没什么关系。这一结果让人十分震惊和失望,因为可能科学家花了20年的时间,加上不计其数的人力和财力,结果研究的可能是根本不存在的东西。同时,这也提示我们仅根据“经验”所提出的猜想可能并不靠谱。换句话说抑郁症候选基因研究所取得的很多成果在今天看来可能只是实验中的偶然,或是由于过度追逐阳性结果所产生的假阳性。

就在研究者为陷入候选基因研究的可重复性危机而苦恼时,随着测序技术的进步,全基因组测序的成本大大降低了。例如2001年进行一个全基因组测序需要花费接近一亿美元,十年后这一价格下降到了一万美元左右,而在今天仅需要不到一千美元就可以完成对一个个体的全基因组测序。

测序成本的降低使得大规模测序成为可能,这使得很多研究者可以不再像以往一样需要依靠“经验”选定单个或者少数候选基因,每次研究也只能围绕单个或者少数基因进行。现在研究者能够在不做理论假设的前提下,对基因组上的数千个基因甚至是整个基因组进行扫描,去寻找疾病相关的致病基因。在这一背景下,全基因组关联分析(GWAS)的时代到来了。
全基因组关联分析(GWAS)
全基因组关联分析不同于以往的候选基因研究,它可以在不做理论假设的前提下,将人群按照疾病特征分成病例组和对照组两个群体,并且借助SNP芯片或者全基因组测序手段对两个群体数以百万计的分子标记(例如SNP和CNV等)进行基因分型,并统计这些分子标记的基因频率在两组人群之间是不是有明显的差异,从而寻找到与复杂疾病发生相关的基因变异。目前,全基因组关联分析(GWAS)已成为人们寻找复杂遗传疾病致病基因的重要手段。据统计,全世界范围内已开展了5500多项GWAS研究,发现了超过三十万个与各种疾病以及重要生理性状关联的基因位点。

抑郁症的致病基因的研究目前也是使用了全基因组关联分析(GWAS))这一策略,但其过程却并不顺利。在抑郁症GWAS研究最开始的几年,由于样本量比较小、样本之间的异质性大等原因,一直没有找到与抑郁症显著相关的基因变异。直到2015年,研究者仅对病情非常严重的5,303例中国汉族女性抑郁症患者与5,337例健康对照样本进行GWAS分析,终于鉴定到两个与抑郁症显著相关的基因变异(rs12415800和rs35936514)。随后的几年通过进一步增大样本量,目前研究者通过GWAS已经鉴定到了接近两百个与抑郁症显著相关的基因变异,这对于研究抑郁症的发病机理具有极大的提示意义。
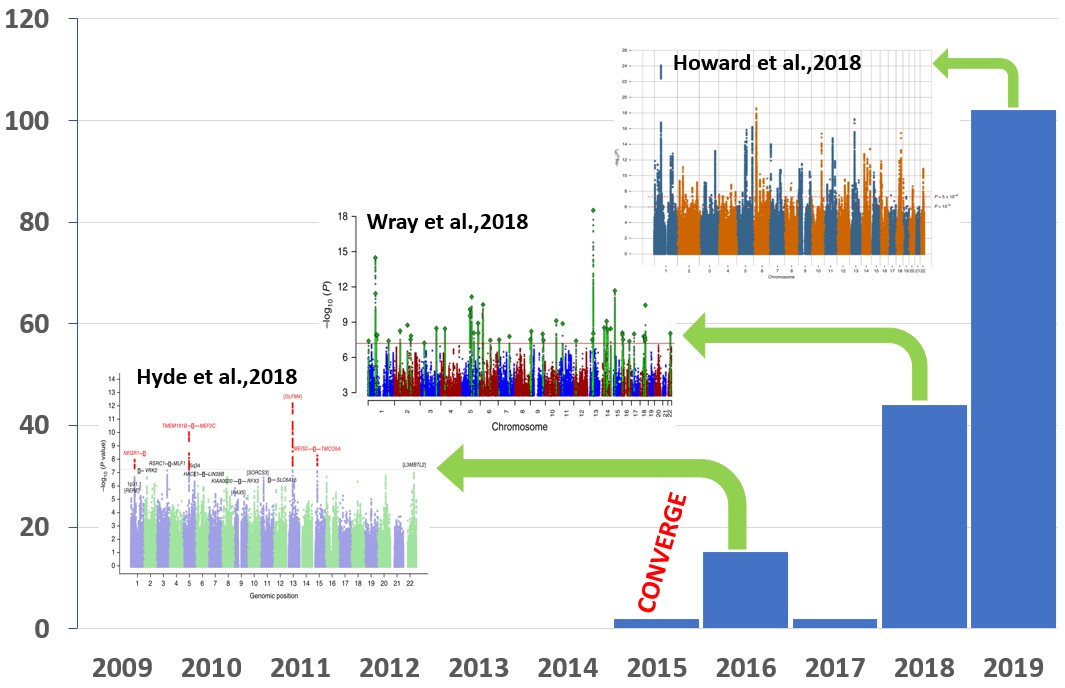
尽管凭借着巨大的样本量和标准化的分析流程,GWAS的研究结果目前表现出了较强的可重复性,说明借助GWAS来寻找与复杂疾病相关的基因变异是一种比较靠谱的方法,但目前的GWAS研究仍然存在一些挑战。
首先目前的GWAS研究对象大部分都是欧美人群,用这些研究结果来解释其他人群(比如中国人)的发病风险可能并不准确,因此,也就要求研究者需要在更多不同种族的人群中开展疾病GWAS研究。
其次,GWAS的结果表明,我们生活中的很多常见疾病的遗传复杂性远超我们目前的认知。这些常见疾病通常并不存在影响特别大的单一遗传变异,而往往是由成百上千的具有微小效应的遗传变异共同作用的结果。目前已经发现的遗传变异的效应也远远达不到双胞胎实验所预测的疾病的遗传力,并由此也就产生了“遗传力缺失”问题,如何解释这一问题将成为GWAS研究中的重要挑战。
最后,如何将越来越多的GWAS信号转化成对发病机制的理解也是目前GWAS研究的最主要挑战。说白了,由于GWAS鉴定到信号仅仅是统计上的相关性,并不能反映出这种关联背后的生物学机制。尤其是目前的研究发现绝大多数(超过98%)的疾病相关遗传变异都位于不能直接编码蛋白的非编码区。可以预见的是,想要搞清楚这些遗传变异的功能将需要大量的时间和人力投入。

参考资料
Sullivan PF, Neale MC, Kendler KS. Genetic epidemiology of major depression: review and meta-analysis. Am J Psychiatry. 2000;157(10):1552-1562.doi:10.1176/appi.ajp.157.10.1552
Border R, Johnson EC, Evans LM, et al. No Support for Historical Candidate Gene or Candidate Gene-by-Interaction Hypotheses for Major Depression Across Multiple Large Samples. Am J Psychiatry. 2019;176(5):376-387. doi:10.1176/appi.ajp.2018.18070881
Wetterstrand KA. DNA Sequencing Costs: Data from the NHGRI Genome Sequencing Program (GSP) Available at: www.genome.gov/sequencingcostsdata.
Tam V, Patel N, Turcotte M, Bossé Y, Paré G, Meyre D. Benefits and limitations of genome-wide association studies. Nat Rev Genet. 2019;20(8):467-484. doi:10.1038/s41576-019-0127-1
Visscher PM, Wray NR, Zhang Q, et al. 10 Years of GWAS Discovery: Biology, Function, and Translation. Am J Hum Genet. 2017;101(1):5-22. doi:10.1016/j.ajhg.2017.06.005







 扫码下载APP
扫码下载APP

 科普中国APP
科普中国APP
 科普中国
科普中国
 科普中国
科普中国